
Les éléments transposables sont des séquences d'ADN mobiles dont l'existence à été mise en évidence par Barbara McClintock dans les années 40 chez le maïs. Ces séquences sont présentes dans toutes les branches de l'arbre du vivant et peuvent représenter une grande partie du génome (environ 40% du génome chez l'Homme et jusqu'à 90% chez le blé). L'universalité et la mobilité de ces séquences accréditent l'idée que les éléments transposables jouent un rôle dans l'évolution et la plasticité des génomes, ce qui est prouvé dans le maïs où un gène impliqué dans la pigmentation est dans certains cas inhibé par un élément transposable qui, lorsque il s'excise, donne un pigment violet au maïs.
En effet, les éléments transposables sont souvent considérés comme égoïstes et ils ont une capacité de réplication à l'intérieur du génome. Les éléments transposables ne sont pas des gènes indispensables à la survie et leur transposition peut causer de grands changements dans le génome. Ces séquences entretiennent des interactions parasitaires avec leur génome hôte, comme le commensalisme ou le mutualisme. Ils peuvent par exemple, parfois, trouver une fonction utile à l'organisme hôte comme l'élément Transib qui a été utilisé pour le système immunitaire adaptatif chez les mammifères. D'autres exemples sont les éléments TART et HetA qui jouent le rôle de la télomérase en ne transposant que dans les télomères d'une espèce de drosophile.
Cette courte introduction ne vous laisse, je l'espère, aucun doute sur l'importance de ces éléments.
Je vais donc tenter de vous donner une méthode pour les trouver, une méthode pour les classer, une méthode pour les analyser et des ténèbres les sortir.
Classifier
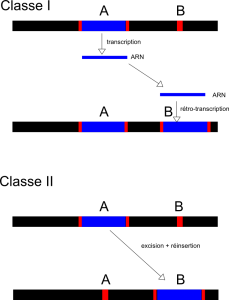
Il existe deux grandes classes d'éléments décrites. Dans les deux classes d'éléments le mécanisme de transposition est très différent.
En effet les éléments de classe I ont un mécanisme de transposition réplicative qui utilise un intermédiaire ARN, laissant la copie originale en place et en ajoutant une autre à un autre emplacement dans le génome selon un mécanisme de "copier-coller" . Les éléments sont transcrits en ARN. Ceux-ci sont alors rétro-transcrits en ADN par une transcriptase inverse. Les ADN sont ensuite réinsérés dans le génome par une intégrase.
Les éléments de classe II codent en général une transposase qui permet un mécanisme de "couper-coller", en excisant un élément présent à un emplacement donné du génome pour l'insérer à un autre. Cette transposase tout comme les protéines des éléments de classe I se base sur la reconnaissance d'une partie de l'élément. Bien que la transposition des éléments de classe II ne soit pas réplicative, il existe des processus qui permettent l'amplification des copies (réparation du site d'excision sur la base du chromosome homologue, synchronisation de la transposition avec la réplication de l'ADN).
Pour en savoir plus sur la classification : « A unified classification system for eukaryotic transposable elements », Wicker et al., Nature Reviews Genetics 8, 973–982 (December 2007) doi:/10.1038/nrg2165-c3
Rechercher
Que ce soit dans un objectif d'étude ou pour les masquer afin d'étudier uniquement les gènes il faut être capable de trouver ces éléments qui, comme nous venons de le voir, sont très différents les uns des autres.
La transposition réplicative augmente rapidement la quantité de copies présentes dans le génome ce qui amène à la principale caractéristique des éléments transposables, le fait qu'ils soient répétés en de nombreuses copies dans le génome.
Deux grands types de méthodes existent donc pour chercher les éléments transposables. La première consiste à utiliser une base de données d'éléments ou de motifs déjà connus (cf paragraphe suivant), ce qui est très rapide mais laisse de coté tout nouvel élément ; la seconde quant à elle consiste à chercher les éléments répétés en de nombreux exemplaires dans le génome et pouvant être associés à une famille, ce qui ne permet de repérer dans un individu que les familles ayant plusieurs copies conservées.
Pour en venir à la bioinformatique, il va de soi qu'il n'est pas possible d'analyser des génomes entiers à la main. Des outils de recherche d'éléments existent donc depuis longtemps avec des logiciels tels que LTR_Struc (2003) qui recherche spécifiquement ou des logiciels plus généralistes utilisant une méthode de novo comme Repet (2011).
Ces logiciels recherchent des motifs et servent donc à l'annotation des éléments transposables présents dans les génomes.
Pour découvrir de façon plus précise la recherche d'éléments répétés dans les génomes je vous conseille la lecture de « Considering transposable element diversification in de novo annotation approaches » Timothée Flutre et al., PLoS ONE 6 (January 31, 2011) doi:10.1371/journal.pone.0016526
Stocker
Aussi bien pour rechercher les éléments transposables déjà analysés que pour stocker les nouveaux éléments trouvés il faut des bases de données. Les éléments transposables étant présents dans tous les génomes il faut une base de données généraliste. Repbase est une base de données gérée par un organisme privé (à but non lucratif) dont le but est le stockage et l'analyse des éléments transposables. Cette base de données est importante pour les gens voulant savoir si les éléments trouvés dans leur génome ressemblent à des éléments connus mais elle est aussi et surtout utilisée par les logiciels de recherche d'éléments.
Si vous avez envie de comprendre comment fonctionne la base de données et les outils associés je vous conseille de lire « Repeats in genomic DNA : mining and meaning. » Jurka, Curr. Opin. Struct. Biol. 8:333–337 (1998). (pubmed)
Masquer

Pour de nombreuses personnes les éléments transposables sont juste des parties gênantes du génomes (que ce soit parce qu'ils représentent une importante masse de données ne codant pas pour des fonctions ou parce qu'à cause de leur aspect répété ils posent problème pour l'assemblage de génomes) et il est donc intéressant de pouvoir les masquer. Des logiciels tels que RepeatMasker permettent ce genre de chose.
Ce logiciel fonctionne en trouvant des zones qui sont répétées dans le génome, ou pour masquer les mini/micro-satellites en recherchant des zones de faible complexité. Une fois ces zones trouvées il retourne un génome masqué en remplaçant les zones de faible intérêt par des X. Le génome résultant se retrouve nettoyé de ses éléments transposables et est plus facile à analyser.
Analyser
Cet article a pour but de faire découvrir des éléments qui, comme décrit en introduction, ont une importance pour expliquer l'évolution des génomes. Des études ont donc été faites sur les éléments transposables.
Que peuvent-ils nous apporter comme information ?
Les éléments transposables sont des facteurs importants de plasticité du génome. Le génome peut être vu comme l'environnement et le prédateur de ces éléments, qu'il tente d'éliminer. Les éléments transposables adoptent donc des fonctionnements assez similaires à ceux retrouvés par les écologistes pour les organismes vivants que ce soit le parasitisme, le mutualisme ou le commensalisme avec des éléments transposables qui s'insèrent dans d'autres éléments. Il est donc possible de faire de nombreuses études sur ces éléments de la même manière qu’elles sont menées sur les organismes vivants, comme l'analyse de liens de parenté ou de survie.
Les éléments transposables peuvent également être étudiés pour leur impact sur le génome comme par exemple dans un article de Bennetzen qui donne un aperçu des éléments transposables comme vecteurs de translocations, de régulation des gènes, d'inactivation. Toutes ces activités ont des conséquences très importantes pour le génome.
Pour une review des connaissances sur les éléments transposables et leur lien avec le génome je vous conseille « The struggle for life of the genome's selfish architects », Hua-Van et al., Biology direct 6, 1745–6150 (2011) (pubmed)
Si vous voulez découvrir les impacts des éléments sur le génome vous pouvez lire « Transposable element contributions to plant gene and genome evolution »,Bennetzen, Plant molecular biology 42, 251–269 (2000) (pubmed)
Un petit plus
Dans les deux classes, on a décrit des éléments non-autonomes qui parasitent une famille d'éléments autonomes associés. Ces éléments transposent mais ne produisent pas leur propre machinerie de transposition. Chez les éléments de classe II il s'agit par exemple des MITEs. Ces éléments parasites utilisent les transposases fournies par ses copies pleines longueur de la famille associée en ayant les mêmes séquences de reconnaissance.
Sur ce sujet je vous conseille « Miniature inverted-repeat transposable elements (MITEs) and their relationship with established DNA transposons », Feschotte et al., Mobile DNA II, 1147–1158 (2002)


Laisser un commentaire