Étiquette : Illumina
-
Pourquoi certains fichiers FASTQ finissent par 001 ?

Sur le chan IRC du blog, un de nos membres se demandait pourquoi les noms de fichiers FASTQ devait finir par _001.fastq sur la plateforme de cloud computing d'Illumina BaseSpace. Mais avant de répondre à cette question pressante, repartons du début. Les fichiers FASTQ En cette période de domination du séquençage haut débit de l'ADN,…
-
Analyse des données de séquençage à ARN
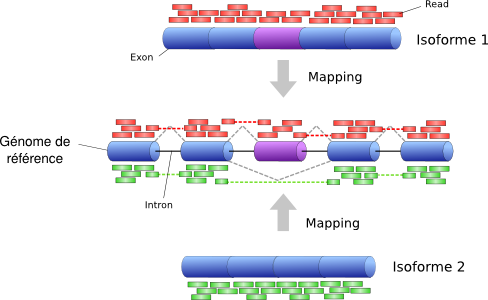
Depuis quelques années, le domaine du séquençage de l'information génétique est rentré dans une nouvelle ère : “le séquençage de seconde génération”. Cette avancée technologique a permis une analyse plus en profondeur de l'ADN et l'ARN. Nous pouvons citer parmi ces nouvelles technologies le ChIP-seq (Chromatine Immuno Precipitation sequencing) ou RNA-seq (Séquençage à ARN). Le projet…


