Qui sommes nous ?
-
Le cauchemar des systèmes de coordonnées en bioinformatique
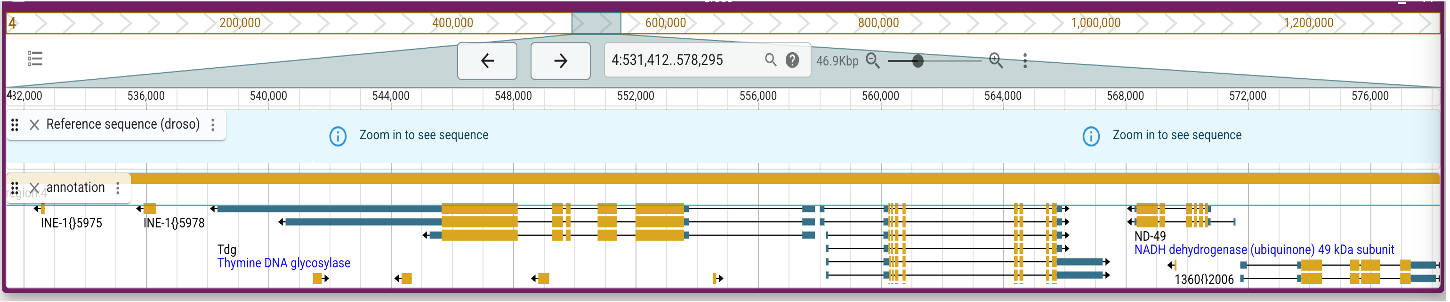 Lire la suite : Le cauchemar des systèmes de coordonnées en bioinformatique
Lire la suite : Le cauchemar des systèmes de coordonnées en bioinformatiqueEn bioinformatique, quand on manipule des génomes, des annotations, des fichiers d’alignement, des variants ou des intervalles (exons, régions régulatrices, etc.), on est rapidement confronté à deux notions fondamentales qui conditionnent la manière dont les positions sont décrites et interprétées : Ces deux dimensions définissent ensemble le système de coordonnées utilisé. Les maîtriser permet d’interpréter correctement […]
-
Trois exemples concrets qui m'ont fait m'intéresser à la reproductibilité entre langages
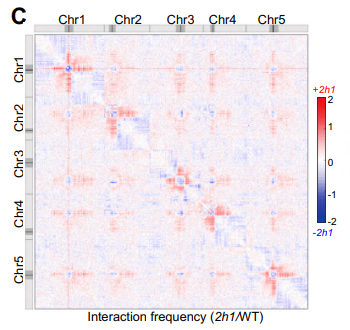 Lire la suite : Trois exemples concrets qui m'ont fait m'intéresser à la reproductibilité entre langages
Lire la suite : Trois exemples concrets qui m'ont fait m'intéresser à la reproductibilité entre langagesMes très chers confrères adorés, Comme vous le savez si vous lisez mes excellents articles d'opinion, tous géniaux (hum !), cela fait quelques années maintenant que je travaille pour des toxicologues. Alors, c'est bien, j'ai appris plein de choses, au point de me définir aujourd'hui comme « computational toxicologist ». Mais j'ai surtout mis mon nez dans […]
-
L'alignement multiple en étoile ⭐
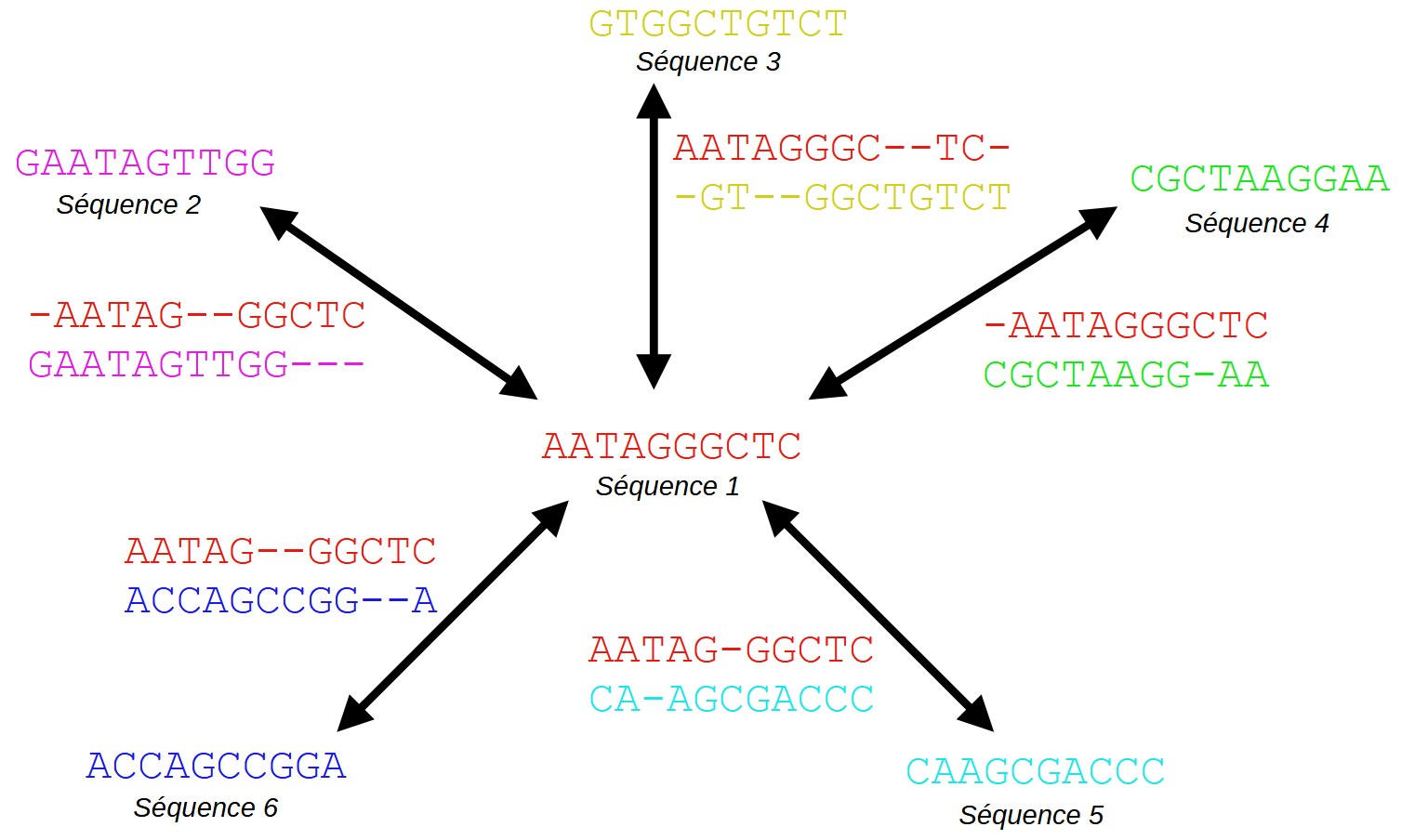 Lire la suite : L'alignement multiple en étoile ⭐
Lire la suite : L'alignement multiple en étoile ⭐Introduction Dans le domaine de la bioinformatique l'alignement de séquences est une opération courante pour comparer des séquences biologiques (ADN, ARN, protéines). Nous avons vu dans un précédent article le principe de l'algorithme de Needleman-Wunsch qui produit un alignement global optimal de deux séquences. Au-delà de l'alignement par paire, l'alignement multiple de séquences (Multiple Sequence […]
-
Bonne rentrée 2025 à tous 🎓
 Lire la suite : Bonne rentrée 2025 à tous 🎓
Lire la suite : Bonne rentrée 2025 à tous 🎓À tous les p'tits veinards qui ont profité de vacances cet été 🌴— et aux autres aussi — on vous souhaite une belle rentrée 🎓. C’est cette période de l’année où l’on prend un nouvel élan : on planifie, on se projette, on fixe de nouveaux objectifs. Peut-être un nouveau poste pour certains, une formation pour […]
-
Licence Bio-Informatique, Statistique et Modélisation (BISM) à Lyon 1
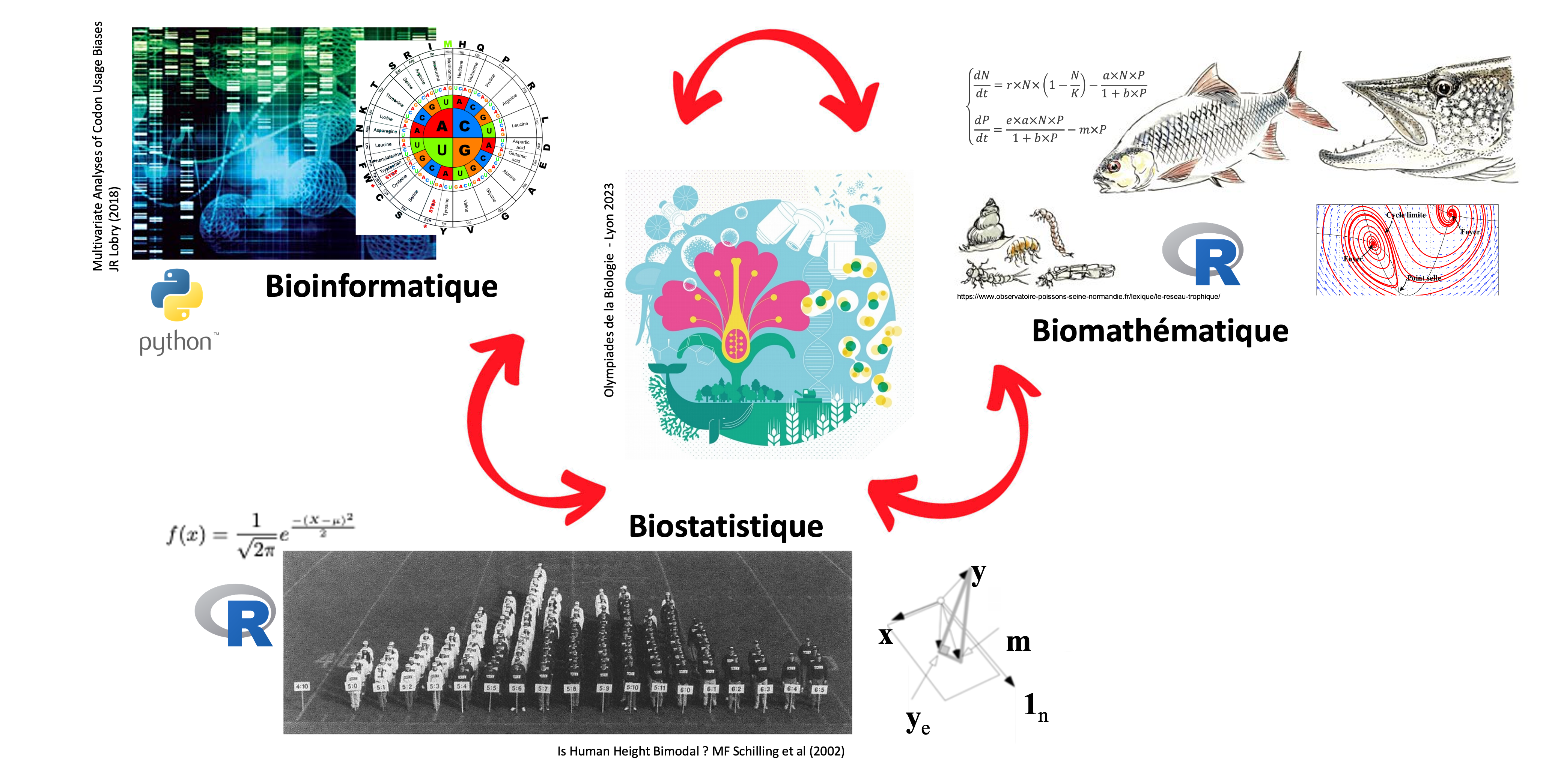 Lire la suite : Licence Bio-Informatique, Statistique et Modélisation (BISM) à Lyon 1
Lire la suite : Licence Bio-Informatique, Statistique et Modélisation (BISM) à Lyon 1Cet article est une mise à jour de https://bioinfo-fr.net/presentation-de-la-licence-miv-a-lyon-1. Cette formation pluridisciplinaire existe depuis 2010 (ex. Modélisation et Informatique du Vivant), et bénéficie de la recherche historique lyonnaise en analyses de données biologiques et notamment en biométrie et statistiques. Les aspects liés à la bioinformatique ont été renforcés à partir de 2018, en lien avec […]
-
Alignement de séquences : bien comprendre l'incontournable algorithme de Needleman-Wunsch
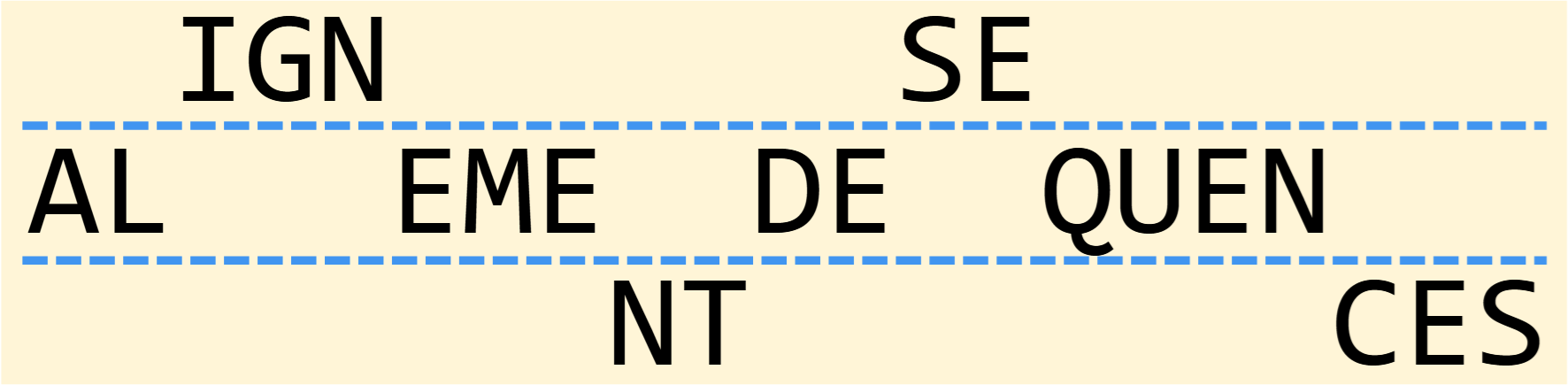 Lire la suite : Alignement de séquences : bien comprendre l'incontournable algorithme de Needleman-Wunsch
Lire la suite : Alignement de séquences : bien comprendre l'incontournable algorithme de Needleman-WunschIntroduction L'analyse des séquences biologiques (ADN, ARN ou protéines) est un sujet central de la biologie moléculaire et de la bioinformatique. Dans ce cadre, chercher à savoir à quel point deux séquences sont similaires ou dissimilaires est une des questions qu'on se pose le plus souvent (par exemple dans le but d'analyser des relations phylogénétiques). […]
-
Des molécules et des données !
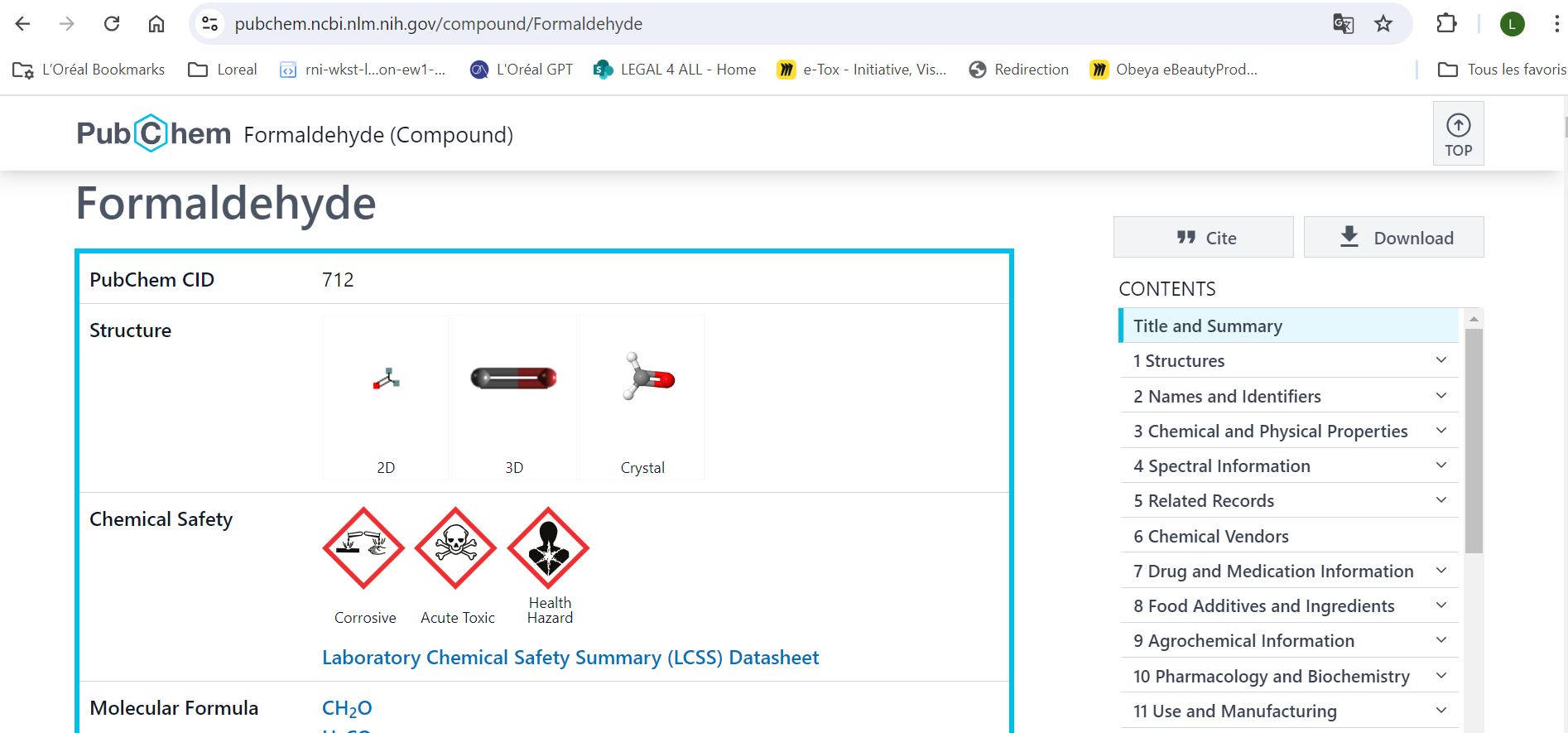 Lire la suite : Des molécules et des données !
Lire la suite : Des molécules et des données !Vu que nous sommes sur un blog de bio-informatique, je vous propose aujourd'hui de parler chimie ! Enfin plutôt, chémoinformatique* et quelques notions de la façon dont une molécule est stockée en ligne en fonction des réglementations chimiques et cosmétiques. Je ne suis pas un expert de ce domaine, mais alors vraiment pas du tout. Par […]
-
Software Heritage : l'archive universelle des codes sources logiciels
 Lire la suite : Software Heritage : l'archive universelle des codes sources logiciels
Lire la suite : Software Heritage : l'archive universelle des codes sources logicielsDans cet article, je vous propose de découvrir Software Heritage, l’archive universelle dédiée à la collecte, la préservation et le partage des codes sources logiciels. En 2021, je suis devenu ambassadeur pour Software Heritage, c’est-à-dire représentant de la communauté bioinformatique, dans l’infrastructure Software Heritage. Pourquoi archiver son code source ? Aujourd’hui, les scientifiques utilisent très souvent des […]
Articles les plus lus ces 7 derniers jours
Les salons de discussion :



La boutique

Quelques liens
Étiquettes
ADN analyse base de données bioinformatique code concours conférence débutant Découverte edito emploi formation Génomique Interview JeBiF JOBIM langage master outil programmation Python R Recherche rentrée script SFBI statistiques séquençage tutoriel vacances visualisation







