Étiquette : graphe
-
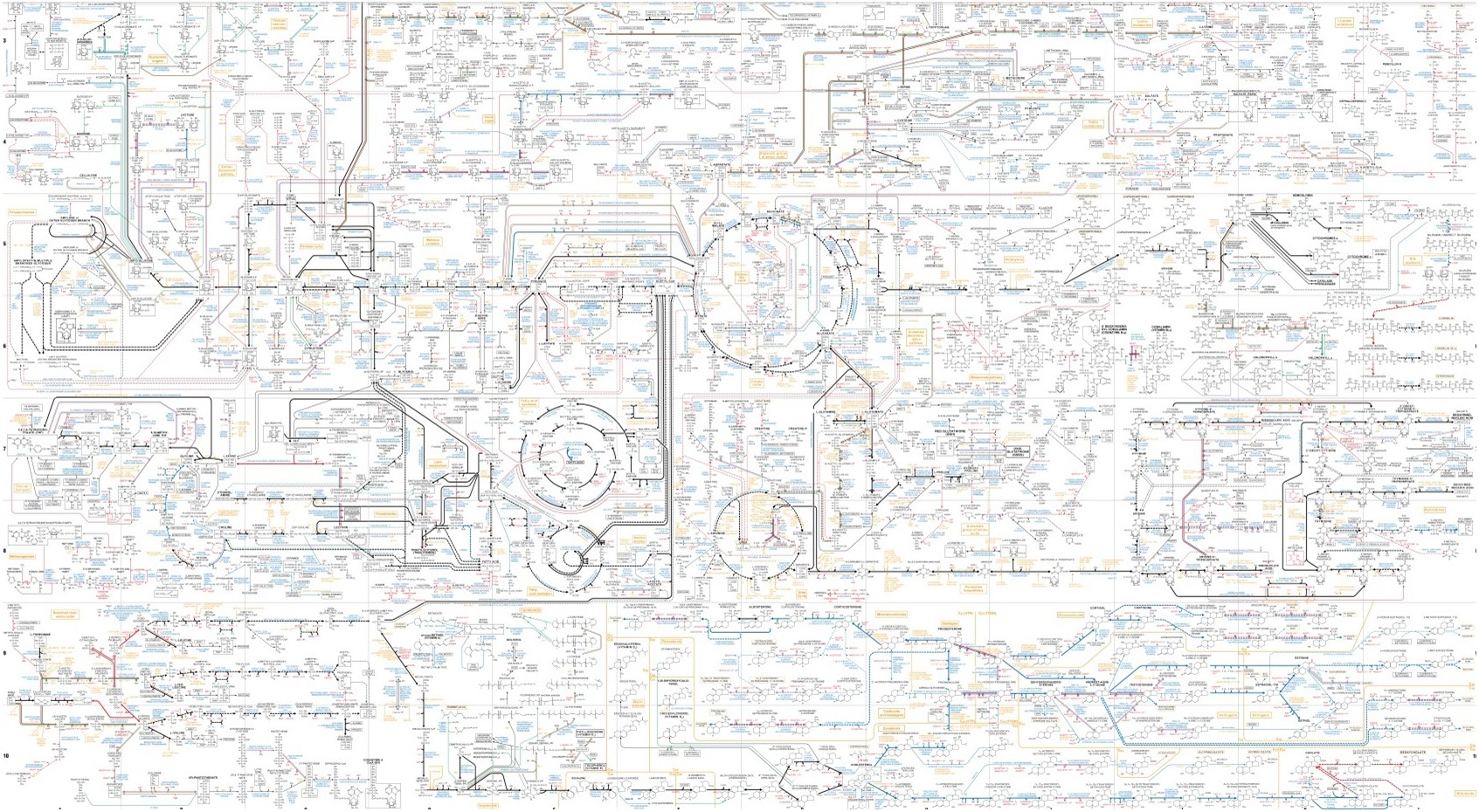
Tour d'horizon des outils de visualisation des réseaux biologiques

Après les différentes méthodes d'analyse et de représentation de réseaux métaboliques biologiques, je vais vous parler des différents outils de visualisation. Car oui, la visualisation d'un réseau ou de ses sous-parties peut être le début de son analyse, car elle permet de se rendre compte de sa topologie, de sa complexité, sa connectivité… En biologie, on peut…
-
Gephi pour la visualisation et l'analyse de graphes

Gephi est un logiciel de visualisation et d'analyse de graphes. Il est distribué sous les licences CDDL 1.0 et GPLv3, et est disponible sur les principaux systèmes d'exploitation. Gephi est prévu d'emblée pour tous types de graphes (pas seulement en bioinformatique) dans les principaux formats. Tulip et Cytoscape sont des outils similaires. Cependant, chez moi…