Catégorie : Didacticiel
-
Chronique d'une soumission de données à GEO

Je vais vous raconter étape par étape ma soumission de données de séquençage à la base de données de génomique GEO (Gene Expression Omnibus) d'un projet en cours de finition. Sommaire GEO, Qu'est-ce que c'est ? Lorsque l'on veut publier les résultats d'une étude comprenant du séquençage haut débit, nous devons publier les données brutes et…
-
R : Convertir des Ensembl IDs en symboles de gènes

Lorsque l'on traite des données de RNA-seq, il arrive très souvent de se retrouver avec une matrice de quantification de l'expression des gènes (un tableau avec le nombre de reads par gène) dont le nom des gènes est représenté par leur identifiant (ou ID) de chez Ensembl (ex. : "ENSG00000128573") et non par leurs symboles…
-
Coder sous Windows avec WSL2

Quand on n'a pas le choix que de coder sous Windows alors qu'on aimerait pouvoir coder sous Unix/Linux, peu d'offres sont disponibles pour développer dans des conditions correctes. Il y en a globalement 2 : soit on crée une machine virtuelle qui fait tourner son OS préféré (ce qui consomme pas mal de ressources du PC…
-
Synchronisez vos PDF Zotero sur Android
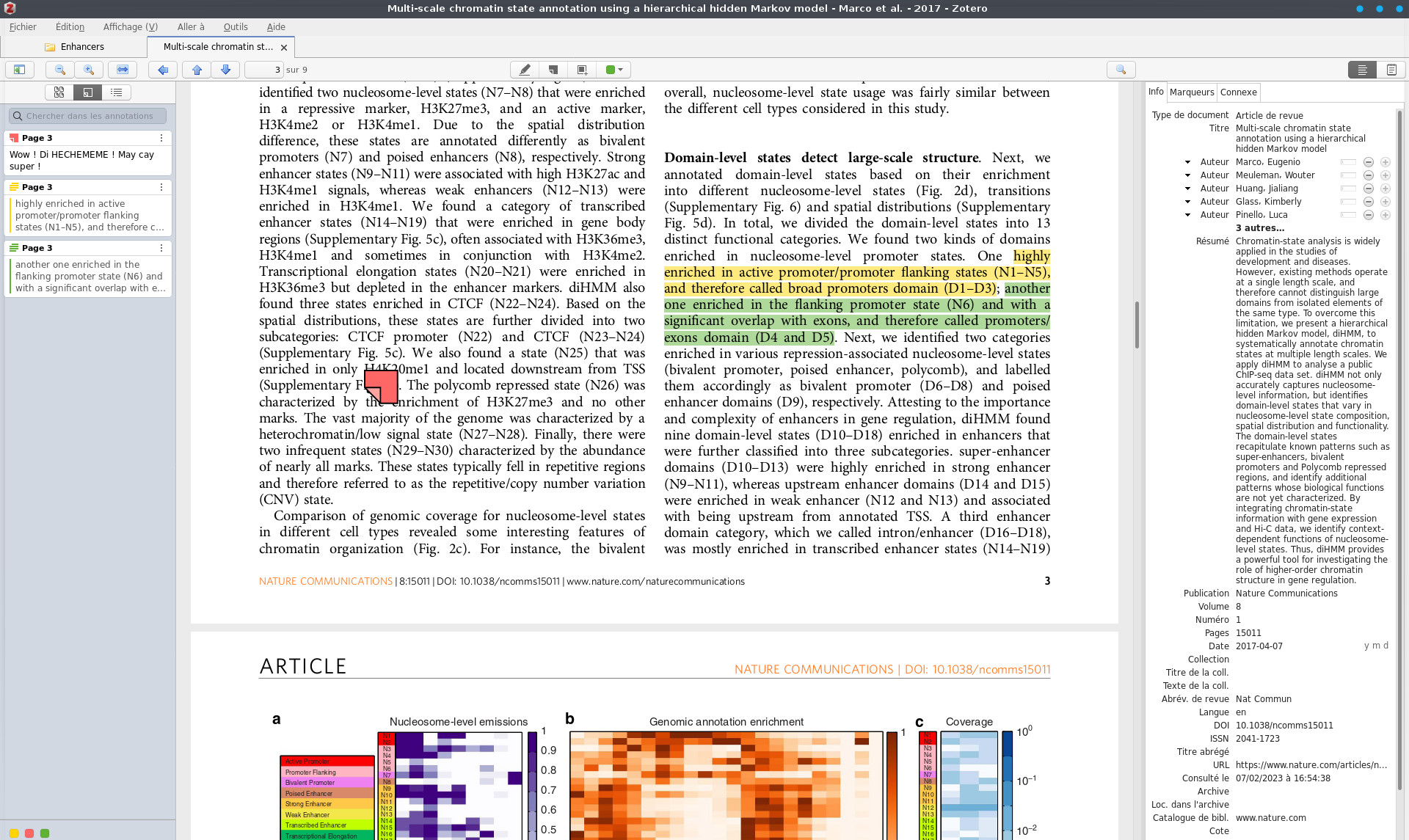
[Update 06/2024] Une application Android officielle est en Bêta Test : https://play.google.com/store/apps/details?id=org.zotero.android&hl=fr Après un an d'errance, j'ai enfin réussi à synchroniser ma collection d'articles en PDF entre mon pc et ma liseuse, et surtout les annotations faites depuis ma liseuse. Bon, j'avais lâché l'affaire pendant longtemps, mais un message de @sebgra sur notre serveur Discord m'a…
-
Créer une documentation automatique avec Doxygen et Sphinx en CI/CD GitLab

Je me suis amusé à écrire une mini librairie C++ pour l'algèbre linéaire (matrices et vecteurs, …) et j'ai eu envie de générer une documentation à partir du code source. Je me suis renseigné un peu et j'ai découvert Doxygen, un outil écrit en C++ qui permet de générer de la documentation à partir des…
-
Installer Overleaf pour des documents LaTeX collaboratifs sur votre serveur
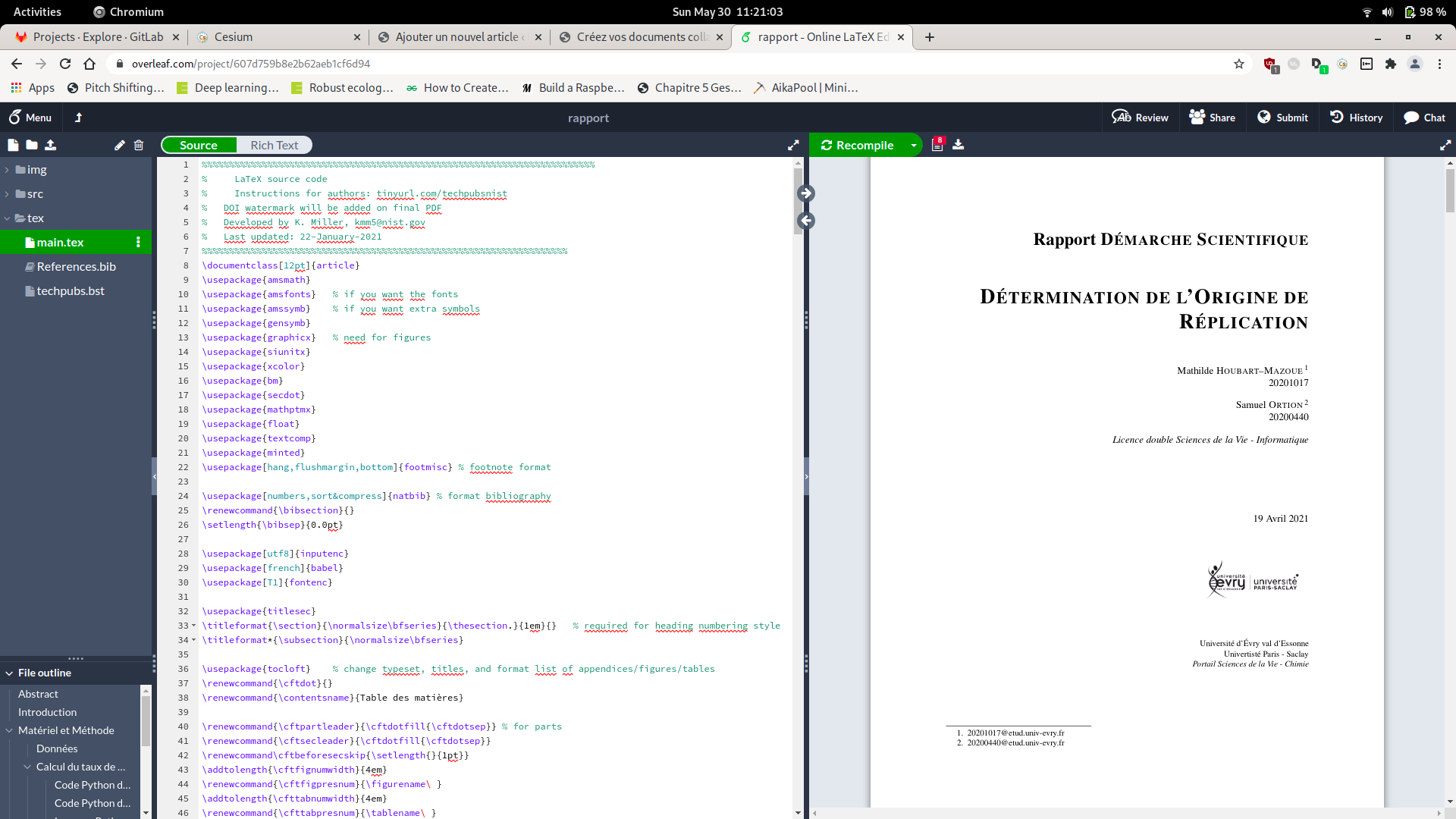
Overleaf est le successeur de ShareLaTeX, il résulte de la fusion de l'ancienne version d'Overleaf avec ShareLaTeX. Il permet d'éditer des documents LaTeX en collaboratif [1]. L'interface se divise en trois parties. Un bandeau pour naviguer dans les fichiers du projet, un éditeur de texte avec coloration syntaxique, et suggestion de commandes LaTeX au fur…
-
Installer JupyterHub pour des Notebooks hébergés sur votre serveur
Vous connaissez sans doute déjà les notebooks Jupyter [1], ces documents web où l'on peut rédiger du contenu en Markdown, pouvant contenir des formules mathématiques en LaTeX, mêlées à des cellules de code Python, (ou R, Julia etc.) que l'on peut exécuter au cas par cas de façon interactive. Ils sont pas mal utilisés en…
-
Organiser une conférence en ligne : trucs et astuces
Organiser une conférence est complexe. Mais organiser une conférence en ligne… c'est TRÈS complexe. Si l'ordinateur qui diffuse la conférence plante… comment prévenir l'audience ? La moindre erreur technique fait d'avantage peur car il devient plus difficile de rebondir et d’expliquer l'issue à la 'salle'. Aujourd'hui je vous propose un partage de toutes les stratégies envisagées pour…

