Étiquette : Métabolisme
-
Promiscuité enzymatique, ou comment gérer l'annotation des enzymes

N'importe quel bioinformaticien, débutant ou confirmé, s'est confronté au moins une fois dans sa vie à ce problème : l'annotation d'une séquence. Vous me direz, faciiiiiile ! Il y a désormais des tonnes d'outils qui permettent, très rapidement en plus, de trouver une fonction pour une séquence ! Alors, déjà, il y a BLAST, InterPro, Pfam, PRIAM, HMMER, ……
-
Représenter le métabolisme - les réseaux métaboliques
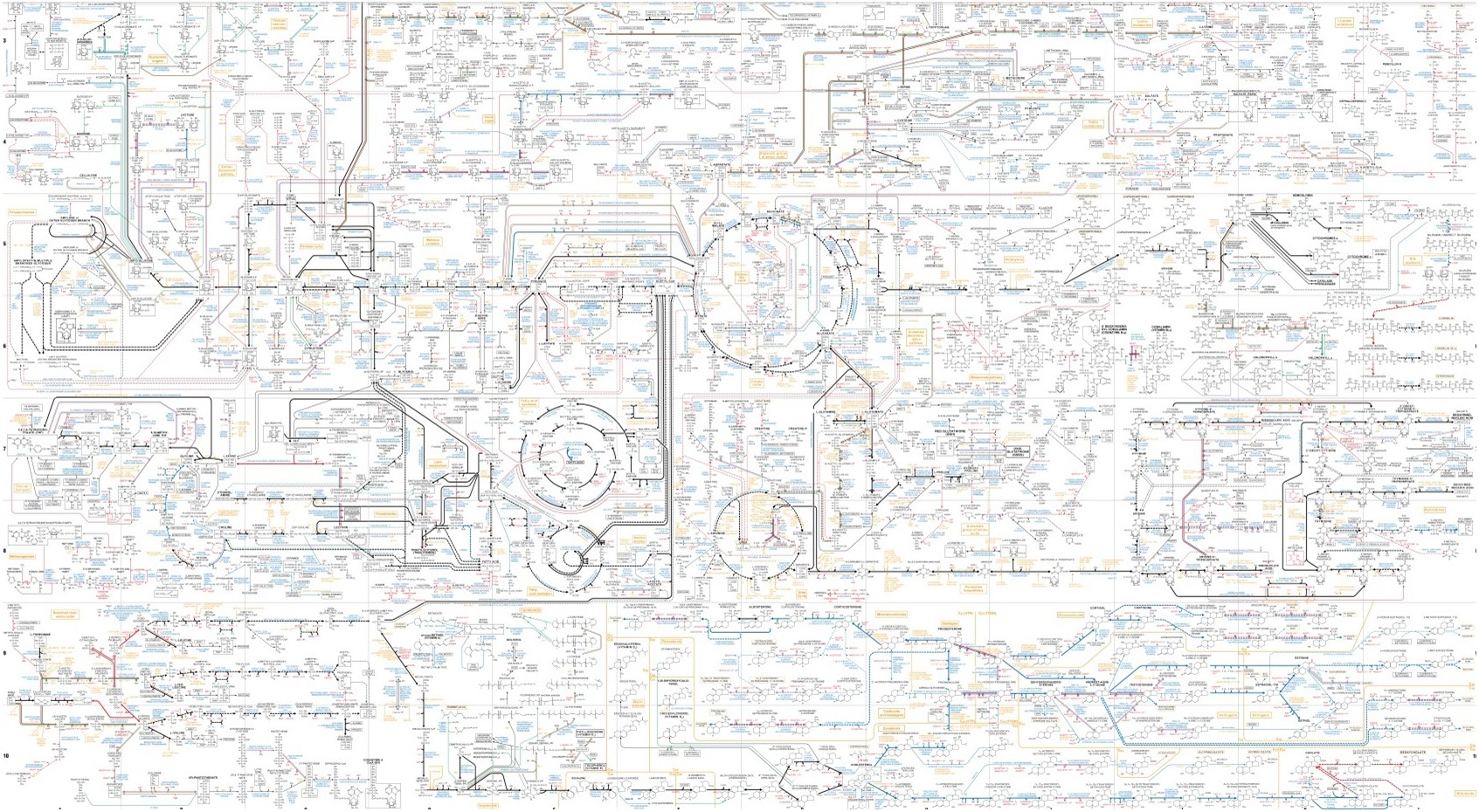
*grosse voix très sérieuse* Attention ! L'article qui suit est le premier d'une série d'articles sur la représentation du métabolisme sous forme de réseaux et leur analyse. Il existe, en bioinformatique, plusieurs catégories de modèles pour décrire le métabolisme.Tout d’abord, les modèles pour l’analyse structurelle du métabolisme. Cette catégorie regroupe principalement les modèles reposant sur la théorie…
-
Un tour d'horizon des bases de données consacrées au métabolisme
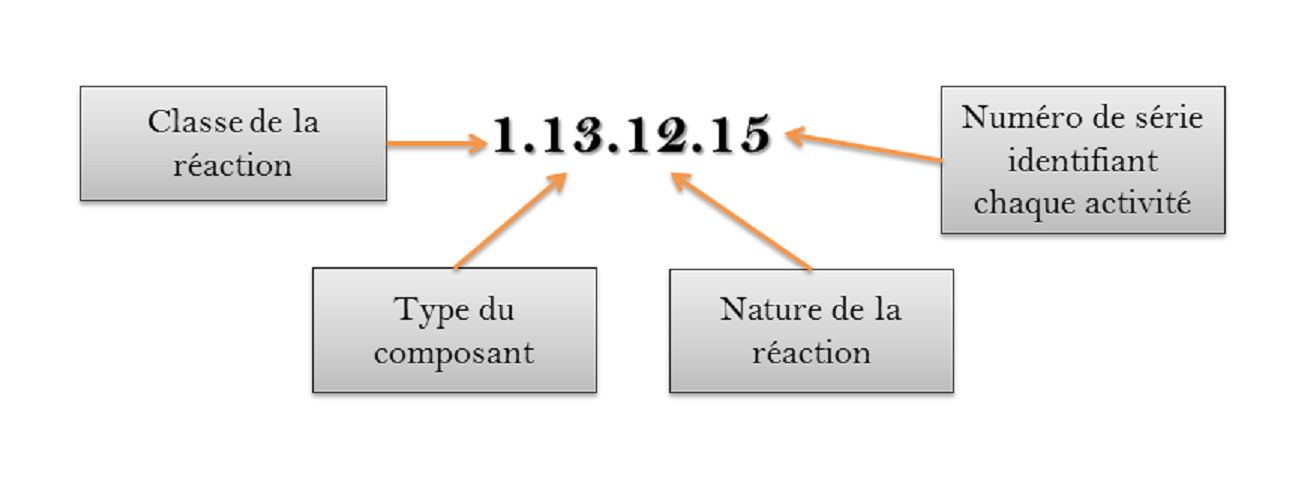
La toute première activité enzymatique a été découverte par Anselme Payen, un chimiste industriel français qui a piqué, grâce à son génie, la domination du marché de borax aux néerlandais. C'était une α-amylase, isolée à partir d'un extrait de malt, et capable de découper l'amidon en glucose. Nommée initialement diastase (synonyme d'"enzyme" à l'heure actuelle),…

