Catégorie : Opinion
-
Les pièges mentaux à éviter pour les plus jeunes chercheurs

Aujourd'hui, j'aimerais exprimer une opinion que je ne vois pas et qui fait suite à quelques conseils données aux doctorants dans ce blog. Ce message s'adresse à tous, dans une communauté à laquelle je n'appartiens plus vraiment n'étant plus dans l'académique. Nous évoquerons ici certains clichés que j'ai entendu. Un jour on est jeune, on…
-
Les IA génératives ne sont pas vos amies !

Cher journal, je t'écris pour répondre à un billet que tu as publié plus tôt. Pour le résumer avec un peu de mauvaise foi : utiliser des IA génératives, c'est trop cool. Contexte ? Je ne vais ici parler que d'IA générative, pas d'autres méthodes d'IA comme celles qui permettent de faire de la classification ou du…
-
Ne devenez pas un dinosaures : adoptez un copilot !

Sauf si vous vivez dans une grotte, sur une planète sans ordinateur ni télévision, le terme ChatGPT doit maintenant résonner à vos oreilles comme le dernier jouet à la mode. Après avoir fait des ravages considérables dans le domaine du traitement du langage, de l'imagerie et de nombreux autres domaines, le Deep Learning est en…
-
Caricature de la bioinformatique pour mon collègue IT
Ce blog n'est pas toujours lisible pour ma mère. Ce blog n'est pas lisible pour mes grands-parents, mais surtout, il ne l'est pas pour toi, mon collègue informaticien. Toi qui ne me comprends pas quand je te dis que je travaille sur des données de grande dimension et qui imagines des images ou du texte.…
-
Pourquoi je ne lis pas tous les CVs de la même manière

Dans un précédent billet je me suis laissé vagabonder autour de l'idée qu'un bioinformaticien était un data truc comme un autre. Dans ces lignes je vais exprimer pourquoi il est important à mon sens de bien travailler son CV… et ce qui personnellement me fait réfléchir sur la forme de CV liée à nos métiers.…
-
De bioinformaticien à data scientist, un simple pas ?
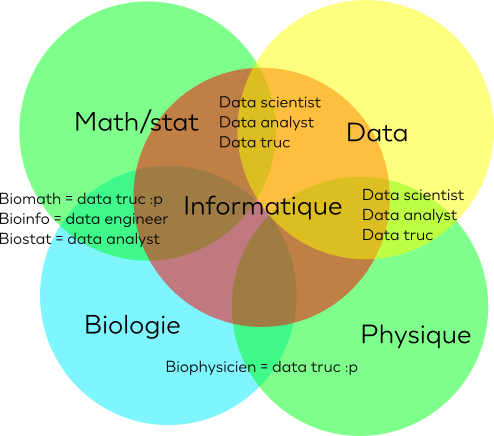
Nombreux parmi nous se retrouvent un jour en fin de master/thèse ou postdoc de bioinformatique. A ce moment là, dans les difficultés de la recherche d'emplois on vient à se demander : "Et si je postulais à toutes ces offres de data scientist ?"Dans ces lignes, je vais donner mon avis de manière libre, sur pourquoi…
-
Qu'est-ce qu'un bon fichier Lisez-moi.txt

Vous venez de finir votre outil sur lequel vous travaillez depuis 1 semaine/1 mois/1 an/10 ans (rayez la mention inutile) qui va révolutionner votre domaine. Votre code est versionné, formaté, commenté, documenté, testé, les résultats sont évalués selon le gold standard de la discipline sur des jeux de données représentatifs de la réalité, votre publication…
-
Choisir entre R et Python en bioinformatique ? Regards croisés entre collègues enseignants-chercheurs

Gaëlle Lelandais et Pierre Poulain Qui sommes-nous ? Tous les deux passionnés par l’enseignement, les problématiques de big data et d’analyse de données en biologie, nous nous côtoyons professionnellement depuis 15 ans, avec écoute et bienveillance. Si l’étiquette de « bioinformaticien » nous est souvent attribuée, nous sommes pourtant très différents. Je (Gaëlle) travaille sur des problématiques de…

