Étiquette : sra
-
Bioconvert - simplifier les conversions de formats
Bioconvert Qui n'a jamais eu à convertir un fichier de données biologiques dans un autre format ? Il y a bien sur le classique fastq vers fasta, pour lequel nombre d'entre nous ont codé un convertisseur "maison", pas forcément optimal. D'autres formats sont parfois plus problématiques, par exemple la conversion vers et depuis GFF2/GFF3. De ces différents…
-
Les bases de données de séquençage : GEO, SRA, ENA, ArrayExpress
De nos jours, lors de la publication de résultats, il est nécessaire de rendre public les éventuelles données de séquençage générées. Si un faible nombre d’irréductibles continuent à ne fournir les données que sur demande, les bonnes pratiques poussent à les déposer dans des bases de données librement accessibles. Quatre grandes bases de données de séquençage…
-
Vous ne savez pas comment analyser vos données Hi-C ? Exemple d'utilisation de HiC-pro
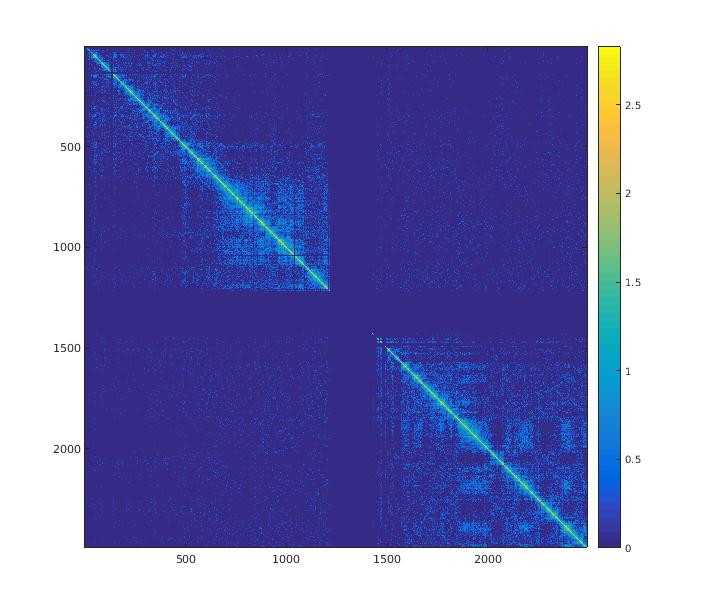
Partir de quelconques données de séquençage haut débit brutes pour arriver à une analyse complète demande au mieux, une certaine pratique de ces technologies. Dans bien des cas, on va alors mettre en place un pipeline reposant sur tout un tas d'outils. Il faudra probablement des heures pour comprendre les paramètres de chacun d’entre eux…

