Comme une partie de la communauté bioinformatique française, et probablement du lectorat de ce blog, je me suis rendu à la 19è édition des Journées Ouvertes en Biologie, Informatique et Mathématiques (JOBIM). Celle-ci se tenait à Marseille, au Palais du Pharo, et pour celles et ceux d'entre vous curieux·ses de connaître le contenu scientifique, voilà le résumé des interventions. Et pour l'ambiance, voilà les photos !
Si vous suivez ce blog depuis longtemps, vous n'êtes pas sans savoir qu'on aime analyser les tweets : c'était le cas pour JOBIM2013, JOBIM/ECCB2014, JOBIM2015… En effet, la communauté bioinformatique francophone gazouille pas mal, et il est possible de récupérer automatiquement les données de ce réseau social. Alors cette année on remet le couvert, mais de manière un peu plus détaillée, et sous forme de réseau ! Et, cerise sur le gâteau, on en fait un tutoriel. On va ici concentrer sur deux mots-dièse (ou hashtag, pour les anglophones) : #JOBIM2018 et #darkjobim (l'existence de ce mot-dièse trollesque est un peu sombre, mais un bioinfo-twittos a une piste).
Alors, dans cet article, nous allons :
- télécharger les tweets des mots-dièse sélectionnés
- les synthétiser sous forme de trois réseaux (i. mots-dièse, ii. twittos et iii. mots-dièse/twittos, un réseau qu'on dit "bipartite")
- interpréter les résultats !
Allez hop, c'est parti ! 💪
Téléchargement des tweets
Pour cette partie, quelques notions de Python seront nécessaires. Si vous n'êtes pas sûr·e d'avoir les bases, n'hésitez pas à fouiller parmi les articles qui concernent ce langage de programmation.
L'idée est d'utiliser l'API de Twitter via le paquet Tweepy. Une API, pour Application Programming Interface, est comme son nom l'indique une interface qui nous permet, dans ce cas précis, d'accéder de manière programmatique au réseau social. Avec Tweepy, on peut tweeter, suivre des gens, faire des recherches… Tout ! Ou presque : l'API de Twitter, ou plutôt sa version gratuite, a quelques limitations, comme l'impossibilité de rechercher des tweets plus vieux que 7 jours à partir de la date actuelle. Mais dans notre cas c'est suffisant : j'ai lancé la requête à la fin de la conférence.
Pour utiliser l'API de Twitter, il faut passer par trois étapes : créer un compte Twitter, demander l'accès Développeur à Twitter, et générer des jetons d'identification. La première étape (obligatoire depuis juillet 2018 seulement, et requérant d'écrire un texte de 300 caractères) se passe ici. Une fois votre compte validé, vous pourrez créer une application, ici, et générer 4 identifiants : les clés d'API (une publique et une secrète) et les jetons d'accès (un public et un secret). Conservez ces 4 identifiants, ce sont eux qui vous permettront de vous connecter à Twitter via Tweepy.
Allez, après toutes ces formalités, mettons un peu les mains dans le cambouis ! Je ne vais pas dans cet article insérer tout le code rédigé pour ce petit projet, mais seulement les éléments les plus importants. Pour les détails et l'implémentation, vous pouvez jeter un œil à l'intégralité du code, disponible en ligne sur Github.
Le premier bout de code important à avoir, c'est donc la connexion à Twitter !
|
1 |
import tweepy<br><br>import json # On va récupérer les tweets au format JSON<br><br># Identifiants obtenus lors de l'enregistrement de l'application<br>consumer_key = "xxx"<br>consumer_secret = "xxx"<br>access_token = "xxx"<br>access_token_secret = "xxx"<br><br># Connexion à l'API<br>auth = tweepy.OAuthHandler(consumer_key, consumer_secret)<br>auth.set_access_token(access_token, access_token_secret)<br>api = tweepy.API(auth) |
# On peut tester en récupérant des tweets de notre propre timeline
public_tweets = api.home_timeline()
for tweet in public_tweets :
print(tweet.text)
Maintenant qu'on est connecté, on va pouvoir faire des requêtes sur les tweets d'un mot-dièse particulier, et les enregistrer dans des dictionnaires. Cela nous permettra de générer les différents réseaux qu'on veut faire à partir d'une seule requête sur Twitter. Pour ceci, la méthode à suivre est relativement simple : elle fait appel à l'objet Cursor de Tweepy, qui permet de récupérer les résultats d'une requête de manière paginée.
|
1 |
# On sauvegarde les tweets dans un dictionnaire<br><br>all_tweets = {}<br><br># On boucle sur les éléments résultant de Cursor<br>for tweet in tweepy.Cursor(api.search, q="#jobim2018").items():<br># On définit un tweet comme un dictionnaire<br>dic = {}<br><br># On peut accéder aux informations du tweet comme ceci :<br>dic['id'] = tweet._json['id']<br>dic['text'] = tweet._json['text']<br>dic['user'] = tweet._json['user']['screen_name']<br>dic['date'] = tweet._json['created_at']<br>dic['fav'] = tweet._json['favorite_count']<br>dic['rt'] = tweet._json['retweet_count']<br><br># Pour les mots-dièse et les mentions, c'est plus compliqué :<br># il peut y en avoir plusieurs, alors on va les stocker sous<br># forme de listes<br>hashtags = tweet._json['entities']['hashtags']<br>hashtags_list = []<br>for h in hashtags:<br>hashtags_list.append(h['text'])<br>dic['hashtags'] = hashtags_list<br><br>mentions = tweet._json['entities']['user_mentions']<br>mentions_list = []<br>for m in mentions:<br>mentions_list.append(m['screen_name'])<br>dic['mentions'] = mentions_list<br><br># On insère dans le dictionnaire des tweets le tweet construit<br>all_tweets[id] = dic |
Ensuite, vu que Twitter ne nous laisse pas accéder à l'intégralité de sa base de données, j'ai enregistré au format JSON cette requête. J'ai ainsi récupéré 669 tweets contenants les mots-dièse #JOBIM2018 et/ou #darkjobim, entre le 1er et le 6 juillet 2018. Le fichier JSON est accessible ici.
Génération des réseaux
Ça y est, on a nos données brutes ! 🎉 Dans cette section, on va les transformer un peu, pour qu'on puisse les visualiser sous forme de réseaux. Pour rappel, on va en construire trois : un réseau de mots-dièse, un réseau des twittos et un réseau qui associe les deux. On pourra donc avoir deux types de nœuds : d'une part les mots-dièse et d'autre part les twittos, qui peuvent être les auteurs des tweets ou bien en mention dans les tweets.
Réseau des mots-dièse
Ce premier réseau ne va donc contenir que des mots-dièse : ce seront les nœuds du réseau. Ils seront connectés par des arêtes, qui n'existeront qu'entre les mots-dièse apparaissant en même temps dans au moins un tweet (notion co-occurrence).
Pour ce faire, on va lire les tweets un par un, et à chaque fois qu'on voit plusieurs mots-dièse dans un même tweet, on met à jour un dictionnaire. S'il s'agit d'un nouveau lien, on crée une entrée dont la clé sera le nom des deux nœuds. Sinon, on met à jour l'entrée déjà existante, qui contient quelques valeurs (nombre de tweets, de retweets, de favoris…). Voilà ce que ça donne en Python :
|
1 |
# On sauvegarde le réseau dans ce dictionnaire<br><br>net = {}<br><br>for id in tweets.keys():<br><br> # On ne garde que la version des mots-dièse en minuscule, pour éviter les doublons<br><br> h = list(map(lambda x:x.lower(), tweets[id]['hashtags']))<br><br># S'il y a plus de 2 mots-dièse, on doit ajouter des liens pour toutes les combinaisons :<br># on calcule ces combinaisons ici<br>comb_h = list(combinations(h,2))<br><br># On trie les mots-dièse par ordre alphabétique pour éviter les doublons<br># ainsi : #a ↔ ; #b et #b ↔ ; #a donneront tous deux : "#a #b"<br>for pair in comb_h:<br># (on ne peut pas changer la valeur d'un tuple, alors on transforme en string pour plus tard)<br>pair0 = pair[0]<br>pair1 = pair[1]<br><br># Parfois un hashtag et un utilisateur ont le même nom, on vérifie ça et on ajoute un suffixe pour différencier<br>if pair0 in unique_mentions:<br>pair0 = pair0 + ' (hashtag)'<br>if pair1 in unique_mentions:<br>pair1 = pair1 + ' (hashtag)'<br><br># Finalement on trie alphabétiquement<br>if (pair1 < ; pair0):<br>entry = pair1 + "\t" + pair0<br>else:<br>entry = pair0 + "\t" + pair1<br><br># On récupère des métriques du tweet, pour pondérer<br>rt = tweets[id]['rt']<br>fav = tweets[id]['fav']<br><br># On enregistre le tout dans un dico de dico<br>if entry not in net.keys():<br>net[entry] = {'n': 1, 'fav': fav, 'rt': rt, 'score': 1+fav+rt}<br>else:<br>net[entry]['n'] += 1<br>net[entry]['fav'] += fav<br>net[entry]['rt'] += rt<br>net[entry]['score'] = net[entry]['n'] + net[entry]['fav'] + net[entry]['rt'] |
Vous l'aurez remarqué : j'enregistre pour chaque association entre deux mots-dièse plusieurs valeurs : le nombre de tweets (n), le nombre de fois que le tweet a été aimé (fav) et retweeté (rt). Je propose également un score (discutable) qui somme le tout. Tout cela va nous servir lors de la visualisation.
On a donc notre réseau de mots-dièse, avec en prime des caractéristiques (appelées "attributs") pour chaque arête. Pour des questions de visualisation, on aimerait aussi avoir des attributs par nœuds, pour savoir combien de fois un mot-dièse a été tweeté par exemple. On fait ça comme ça :
|
1 |
# On sauvegarde les attributs des nœuds dans un… dictionnaire, oui encore !<br><br>attr = {}<br><br>for id in tweets.keys():<br><br> # On ne garde que la version des mots-dièse en minuscule, pour éviter les doublons<br><br> h = list(map(lambda x:x.lower(), tweets[id]['hashtags']))<br><br># … et les utilisateurs et mentions dans les tweets<br>m = tweets[id]['mentions'] + [tweets[id]['user']]<br>m = list(map(lambda x:x.lower(), m))<br><br># Pour chaque mot-dièse, on crée ou met à jour une entrée<br>for h2 in h:<br># Parfois un hashtag et un utilisateur ont le même nom, on vérifie ça et on ajoute un suffixe pour différencier<br>if h2 in unique_mentions:<br>h2 = h2 + ' (hashtag)'<br><br>rt = tweets[id]['rt']<br>fav = tweets[id]['fav']<br><br># Nouvelle entrée<br>if h2 not in attr.keys():<br>attr[h2] = {'type': 'hashtag', 'tweets': 1, 'fav': fav, 'rt': rt, 'score': 1*(fav+rt)}<br># Mise à jour d'une entrée<br>else:<br>attr[h2]['tweets'] += 1<br>attr[h2]['fav'] += fav<br>attr[h2]['rt'] += rt<br>attr[h2]['score'] = attr[h2]['mentions'] * (attr[h2]['fav'] + attr[h2]['rt'])<br><br># Même chose pour les twittos<br>for m2 in m:<br>if m2 in unique_hashtags:<br>m2 = m2 + ' (user)'<br><br>rt = tweets[id]['rt']<br>fav = tweets[id]['fav']<br><br>if m2 not in attr.keys():<br>attr[m2] = {'type': 'user', 'tweets': 1, 'fav': fav, 'rt': rt, 'score': 1*(fav+rt)}<br>else:<br>attr[m2]['mentions'] += 1<br>attr[m2]['fav'] += fav<br>attr[m2]['rt'] += rt<br>attr[m2]['score'] = attr[m2]['tweets'] + attr[m2]['fav'] + attr[m2]['rt'] |
Le code pour générer les deux autres réseaux sont très similaires, vous pouvez jeter un œil dans le code en ligne (fonctionstweets_to_mentions() et tweets_to_bipartite()).
Écriture du fichier
Nous avons à présent nos jolis dictionnaires Python qui rassemblent nos nœuds et nos arêtes, représentant nos différents réseaux. Et maintenant ? Pour visualiser tout ça, il faut convertir cette information en un format que votre logiciel favori supporte. Dans mon cas, j'ai choisi Gephi, notamment parce qu'un super article (encore un !) parle de lui sur ce blog.
Note : d'autres logiciels sont évidemment disponibles, je vous encourage à lire un article, tout aussi génial, qui fait le tour d'horizon des outils de visualisation des réseaux biologiques.
De plus, le code disponible en ligne supporte également l'export au format Cytoscape.
On va utiliser le format GDF, qui ressemble à ça :
|
1 2 3 4 5 6 7 8 9 |
nodedef> ; name VARCHAR, score DOUBLE<br> a,1<br> b,2<br> c,1<br> d,9<br> edgedef> ; node1 VARCHAR, node2 VARCHAR, color VARCHAR<br> a,b,blue<br> b,c,blue<br> b,d,red |
Une première partie ( nodedef ) définit les nœuds ; il est possible d'ajouter autant d'attributs qu'on veut. S'ensuit la partie définissant les arêtes ( edgedef ), avec également la possibilité d'ajouter des attributs.
Dans un premier temps, on va donc lister les nœuds présents et leurs attributs, puis les arêtes :
|
1 |
# On ouvre notre fichier d'export<br><br>f = open("jobim2018_hashtags.gdf", 'w')<br><br>### NŒUDS ###<br>#############<br># On charge ici le nom des attributs, sous forme de liste<br>col_attributes = attr[list(attr)[0]]<br>col_attributes_list = list(col_attributes.keys())<br><br># On écrit l'en-tête du fichier, qui contient le nom des attributs,<br># avec le type de variable auquel ils correspondent<br>f.write('nodedef>')<br>f.write('name VARCHAR')<br>for col in col_attributes_list:<br>if isinstance(col_attributes[col], str):<br>col_type = "VARCHAR"<br>elif isinstance(col_attributes[col], int):<br>col_type = "DOUBLE"<br>else:<br>col_type = "VARCHAR"<br>f.write(', ' + col + ' ' + col_type)<br><br># Les attributs sont générés pour tous les tweets, et si on ne veut<br># que les mots-dièse, on aura aussi des attributs pour les twittos.<br># Solution : on ne garde que les nœuds présents dans le réseau. Ici<br># on liste tous les nœuds présents<br>present_nodes = list(map(lambda key : key.split("\t"), net.keys()))<br>present_nodes = [item for sublist in present_nodes for item in sublist] # Ça c'est pour désimbriquer les listes<br>present_nodes = set(present_nodes) # Et ça pour ne garder que les éléments uniques<br><br># Finalement on écrit les nœuds et leurs attributs<br>for node in attr.keys():<br># On vérifie que le nœud est dans le réseau<br>if node in present_nodes:<br><br># On écrit le nom du nœud<br>f.write('\n' + node)<br><br># Et on boucle sur ses attributs<br>for col in col_attributes_list:<br>f.write(',' + str(attributes[node][col]))<br><br>### ARÊTES ###<br>##############<br># On charge ici le nom des attributs, sous forme de liste<br>col_edges = net[list(net)[0]]<br>col_edges_list = list(col_edges.keys())<br><br># On écrit "l'en-tête" de la deuxième partie de fichier<br>f.write('\nedgedef>')<br>f.write('node1 VARCHAR, node2 VARCHAR')<br>for col in col_edges_list:<br>if isinstance(col_edges[col], str):<br>col_type = "VARCHAR"<br>elif isinstance(col_edges[col], int):<br>col_type = "DOUBLE"<br>else:<br>col_type = "VARCHAR"<br>f.write(', ' + col + ' ' + col_type)<br><br># On écrit les arêtes et leurs attributs<br>for edge in net.keys():<br>f.write('\n' + edge.replace('\t', ','))<br>for col in col_edges_list:<br>f.write(',' + str(net[edge][col]))<br>f.write('\n')<br>f.close() |
Ayé, après ça vous devriez avoir un super fichier de réseau, lisible par Gephi ! Il ne vous reste plus qu'à l'ouvrir pour le visualiser. 😎 N'oubliez pas le didacticiel Gephi sur le blog si vous êtes perdu·e.
Résultats et discussion
Le réseau des mots-dièse
Voilà donc à quoi ressemble notre réseau final :
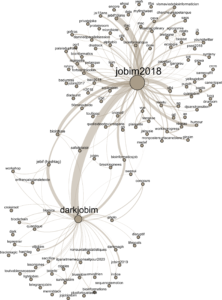
Ici la taille des nœuds représente le nombre de tweets comportant le mot-dièse, mais visuellement les autres métriques donnent un résultat très similaire. On voit donc deux gros paquets, autour des deux mots-dièse qu'on a sélectionné.
Côté #darkjobim, les discussions semblaient plutôt légères : ça parlait cigales, blackmagic, disruptif, jojobimbim et blockchain quantique. Quelques tweets associaient #darkjobim et #jobim2018, à propos de Bioinfuse, bioinformaticsjob, jebif…
Bon, et pour #jobim2018 alors ? Eh bien d'après les mots-dièse, une partie des tweets étaient sérieux (synteny, rstats, microbiote, sfbi, AG). Pour le reste… voyez par vous-même. 🤐
Le réseau des twittos
Concernant les interactions entre twittos eux-mêmes, ça part nettement plus dans tous les sens :

On voit que certains twittos ont été assez prolifiques (je ne donnerai pas de noms). Le réseau est formé d'un grand module interconnecté (on appelle ça une composante connexe), et d'autres plus petites avec deux ou trois twittos seulement. Et en un coup d'œil à ce réseau, on note que le hub Pasteur porte également bien son nom ! 😬
N'hésitez pas à télécharger les fichiers Gephi et les ouvrir avec le logiciel pour naviguer facilement dans le réseau.
Bon, et qu'est-ce que les twittos se sont racontés alors ? On va le voir avec le prochain réseau !
Le réseau bipartite : mots-dièse — twittos
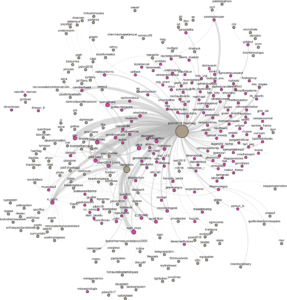
Dans ce réseau on a deux types de nœuds (mots-dièse et twittos), et on ne peut pas connecter deux nœuds d'un même type. Par conséquent, ce qu'on voit c'est littéralement "qui a dit quoi" ! Beaucoup de twittos n'ont utilisé que le mot-dièse #jobim2018 (le groupe de nœuds vers la droite du réseau). Et puis d'autres ont visiblement eu plus d'imagination. 🗣
Conclusion
Pfiou, on aura vu pas mal de choses dans cet article mine de rien ! Comment récupérer des tweets et les enregistrer dans un fichier JSON, comment à partir de ces tweets établir différents réseaux, et comment les visualiser. Et avec tout ça on aura eu un petit aperçu de JOBIM2018 ! Les techniques utilisées, surtout pour générer et visualiser les réseaux, peuvent être adaptables pour étudier des réseaux biologiques (interaction protéine-protéine, similarité de séquences…)
Pour ce petit projet, des améliorations sont possibles, en voici quelques-unes (n'hésitez pas à en ajouter dans les commentaires) :
- un petit nombre de mots-dièse (ou twittos) concentrent beaucoup de tweets, ce qui rend l'échelle de la taille des nœuds assez peu représentative (on ne voit pas la différence entre 1 et 20 tweets). On pourrait changer cette échelle.
- il y a d'autres mots intéressants dans les tweets que les mots-dièse, on pourrait aussi les utiliser.
Je rappelle que le code que j'ai écrit est en ligne à cette adresse : https://github.com/bioinfo-fr/hashtag-network
Enfin, petit coup de pub à une école d'été à laquelle j'ai pu participer sans laquelle rien n'aurait été possible grâce à laquelle j'ai appris à construire des réseaux : Evolunet. 🤓
Encore une fois, énorme merci aux relecteurs et admins (en l'occurrence Mathurin & Yoann M.) ! 💜
Alors, arriverez-vous à reproduire tout ça lors la prochaine conférence à laquelle vous participerez ? 😃


Laisser un commentaire