Étiquette : Python
-
Choisir entre R et Python en bioinformatique ? Regards croisés entre collègues enseignants-chercheurs

Gaëlle Lelandais et Pierre Poulain Qui sommes-nous ? Tous les deux passionnés par l’enseignement, les problématiques de big data et d’analyse de données en biologie, nous nous côtoyons professionnellement depuis 15 ans, avec écoute et bienveillance. Si l’étiquette de « bioinformaticien » nous est souvent attribuée, nous sommes pourtant très différents. Je (Gaëlle) travaille sur des problématiques de…
-
Installer JupyterHub pour des Notebooks hébergés sur votre serveur
Vous connaissez sans doute déjà les notebooks Jupyter [1], ces documents web où l'on peut rédiger du contenu en Markdown, pouvant contenir des formules mathématiques en LaTeX, mêlées à des cellules de code Python, (ou R, Julia etc.) que l'on peut exécuter au cas par cas de façon interactive. Ils sont pas mal utilisés en…
-
J'ai lu : Programmation en Python pour les sciences de la vie

Python et la bioinformatique associés dans un livre, qui plus est écrit par deux maîtres de conférences passionnés de l'Université Paris Diderot que sont Patrick Fuchs et Pierre Poulain : nous nous devions de nous procurer cet ouvrage et de vous en faire une rapide retranscription ! Ce bouquin date de juillet 2019 et c'est tout simplement…
-
Concours : Noël avant l'heure !

A l'occasion de la sortie de notre fiche de lecture sur le livre "Programmation en Python pour les sciences de la vie" et sur une gentille proposition de Pierre Poulain & Patrick Fuchs, nous vous proposons un petit jeu concours avant Noël. Pour participer : il y a dans le livre un petit easter egg placé…
-
Rendre un pipeline Snakemake à l'épreuve des plateformes

Pour avoir été client des articles ("Snakemake pour les nuls", "Formaliser ses protocoles avec Snakemake" et "Snakemake, aller plus loin avec la parallélisation") de mon prédécesseur lelouar, j'ai décidé d'apporter ma pierre à l'édifice et de continuer cette série sur Snakemake. Je vais ici vous parler de généralisation de pipeline pour l'utilisation intensive au sein…
-
Les problèmes limités par les entrées/sorties (IObound)
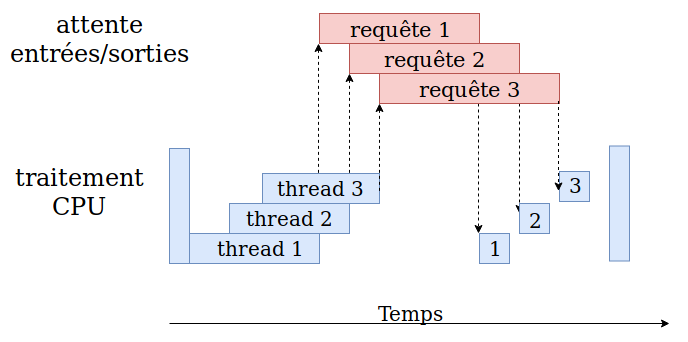
Dans la première partie de ce tutoriel , j'ai expliqué ce qu’était la programmation concurrente et parallèle, ainsi que détaillé les différents types de programmation concurrente et leurs spécificités. Si vous ne l'avez pas lue, je vous conseille de la lire avant de démarrer. Dans cette deuxième partie, nous allons nous concentrer sur l'optimisation d'un…
-
La programmation concurrente en python

Ce tutoriel est une traduction infidèle d'un article de realpython.com https://realpython.com/python-concurrency/#when-to-use-concurrency Merci à eux pour leur formidable travail et leur autorisation. Vous avez certainement entendu parler de la librairie asyncio qui a été ajouté à Python 3 et vous êtes curieux de savoir comment elle se place par rapport aux autres méthodes de programmations concurrentes ?…
-
ViLoVar : un outil pour la visualisation de variations génétiques
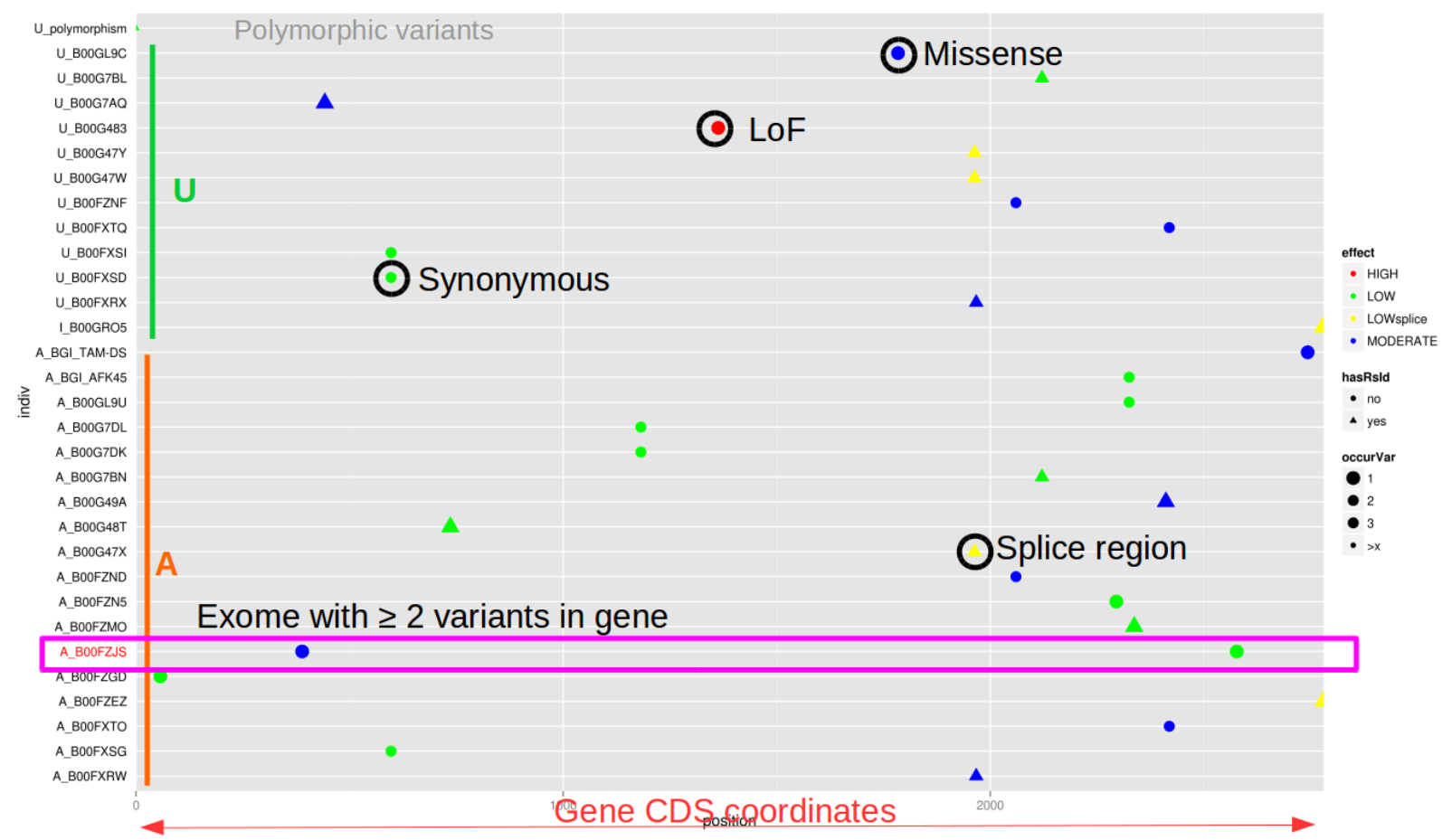
Pour mon premier article, je vais vous présenter un outil que j'ai développé lorsque je travaillais sur le projet "Myocapture"; un projet national de séquençage d'exomes qui portait sur les myopathies (https://www.afm-telethon.fr/myopathie-congenitale-6675). Ce projet visait à trouver de nouvelles mutations responsables de ces maladies rares. Il a également permis d'identifier de nouveaux gènes impliqués dans…

