Étiquette : annotation
-
Qu'est-ce qu'un site d'initiation de la transcription ?
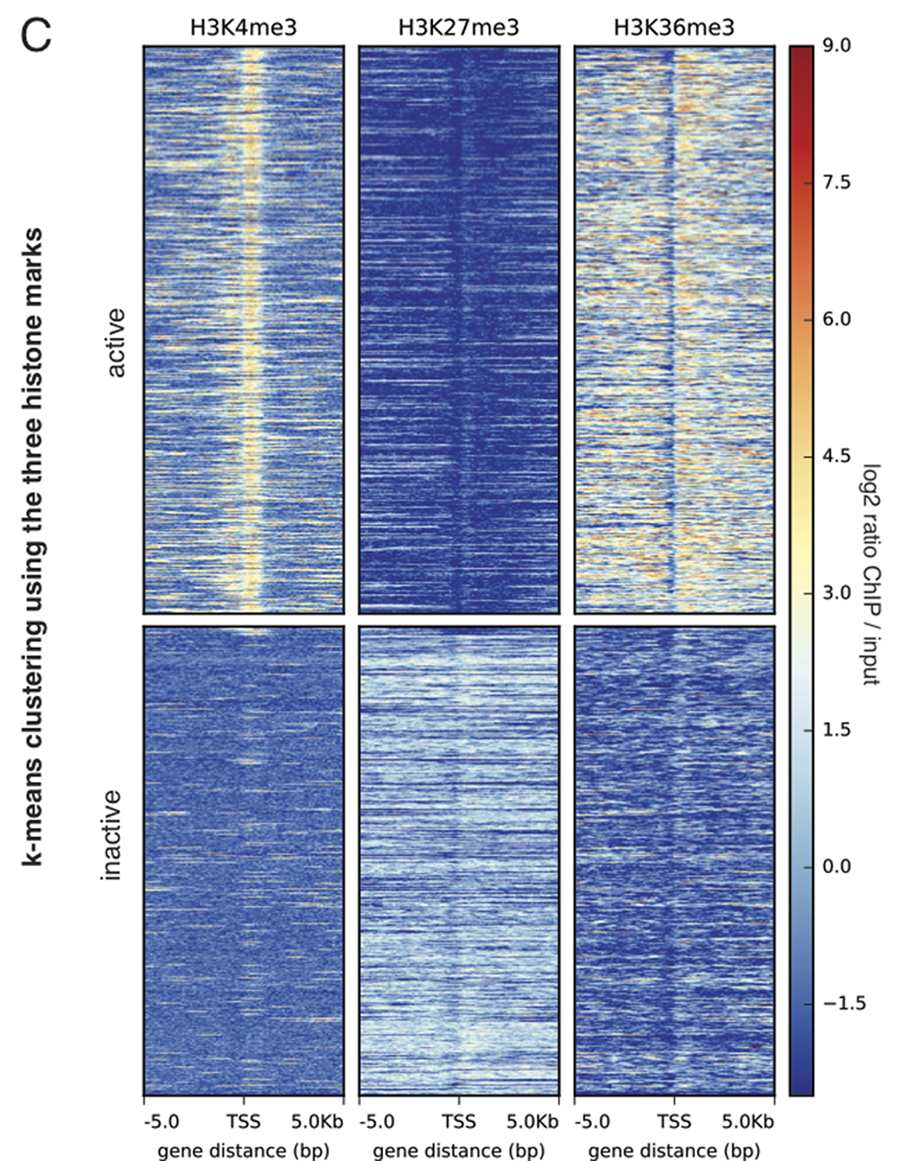
Comment je me suis posé la question. Chez les eucaryotes, l'ADN est organisé en domaines plus ou moins compactés, avec des taux de transcription plus ou moins élevés, et qui sont marqués différentiellement par un certain nombre de marques épigénétiques (méthylation de l'ADN, modifications post-traductionnelles des histones, variants d'histones, etc.). Il est fréquent d'essayer de…
-
Les éléments répétés du génome humain : aperçu rapide avec R et le tidyverse
Dans un précédent article, nous avions regardé le fichier d'annotation des gènes du génome humain d’après Gencode. J'avais utilisé pour cela la puissante combinaison dplyr + ggplot2 (packages centraux du tidyverse), particulièrement adaptée à tout ce qui est manipulation et visualisation de données tabulaires. Mais notre génome n'est pas constitué que de gènes, loin s'en…



