Étiquette : workflow
-
Formaliser ses protocoles avec Snakemake
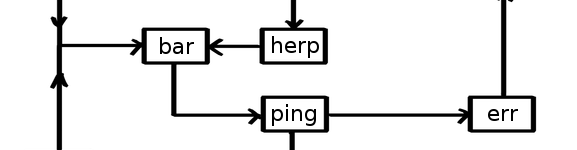
Vous avez dit protocole ? Qui dit biologie et bioinformatique dit protocole expérimental. C'est le cœur de la démarche scientifique, et un formalisme adapté est la clef pour assurer la reproductibilité des expériences, et ainsi garantir la validation des découvertes par la communauté. En paillasse, les solutions pour formaliser et conserver les protocoles sont plutôt naturellement…
-
Introduction aux pipelines
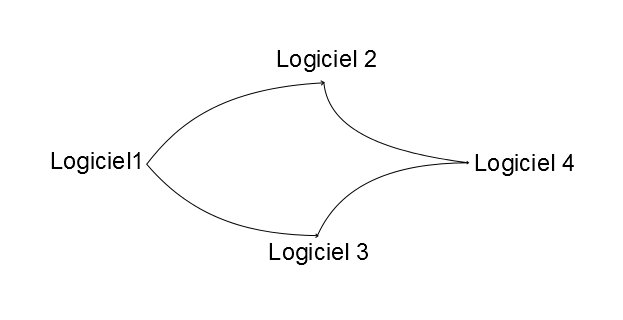
Il vous est peut-être arrivé d'attendre qu'un logiciel A ait fini son travail pour pouvoir lancer un logiciel B, qui lui utilise la sortie du logiciel A. Si l'exécution de A ne prend que quelques secondes cela n'est pas trop grave. Par contre, si elle prend des heures et que vous devez vérifier régulièrement s’il…
-
Galaxy : Bien plus qu'un gestionnaire de workflows

Qu'est-ce que Galaxy ? Galaxy est une application web écrite en Python destinée à faciliter la manipulation et l'analyse des données, dans le cadre de la recherche biomédicale. Elle permet d'utiliser des logiciels habituellement exécutés en ligne de commande de manière graphique, grâce à un système de plugins (« outils ») en XML et de templates Mako. Ces…

