Catégorie : Didacticiel
-
Pourquoi et comment déposer un package R sur Bioconductor ?

Ça y est, votre code R un poil brut commence à avoir de la substance et vous envisagez d'en faire un outil à part entière. Comme tout bioinformaticien qui se respecte, vous envisagez donc de packager (ou paqueter en français) proprement cet ensemble de scripts R. Non on ne largue pas une nuée de scripts…
-
Rendre un pipeline Snakemake à l'épreuve des plateformes

Pour avoir été client des articles ("Snakemake pour les nuls", "Formaliser ses protocoles avec Snakemake" et "Snakemake, aller plus loin avec la parallélisation") de mon prédécesseur lelouar, j'ai décidé d'apporter ma pierre à l'édifice et de continuer cette série sur Snakemake. Je vais ici vous parler de généralisation de pipeline pour l'utilisation intensive au sein…
-
Les problèmes limités par les entrées/sorties (IObound)
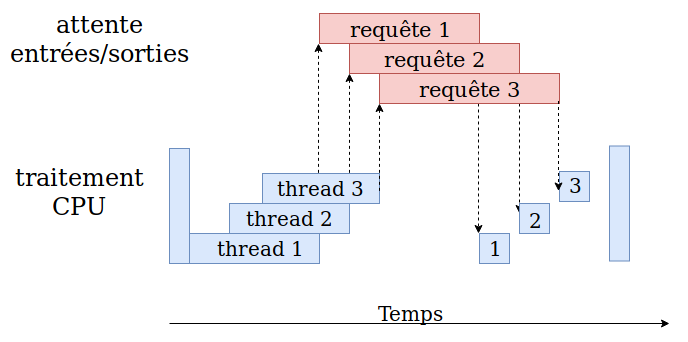
Dans la première partie de ce tutoriel , j'ai expliqué ce qu’était la programmation concurrente et parallèle, ainsi que détaillé les différents types de programmation concurrente et leurs spécificités. Si vous ne l'avez pas lue, je vous conseille de la lire avant de démarrer. Dans cette deuxième partie, nous allons nous concentrer sur l'optimisation d'un…
-
Télécharger des données de séquençage sur le NCBI.. pour les débutants !
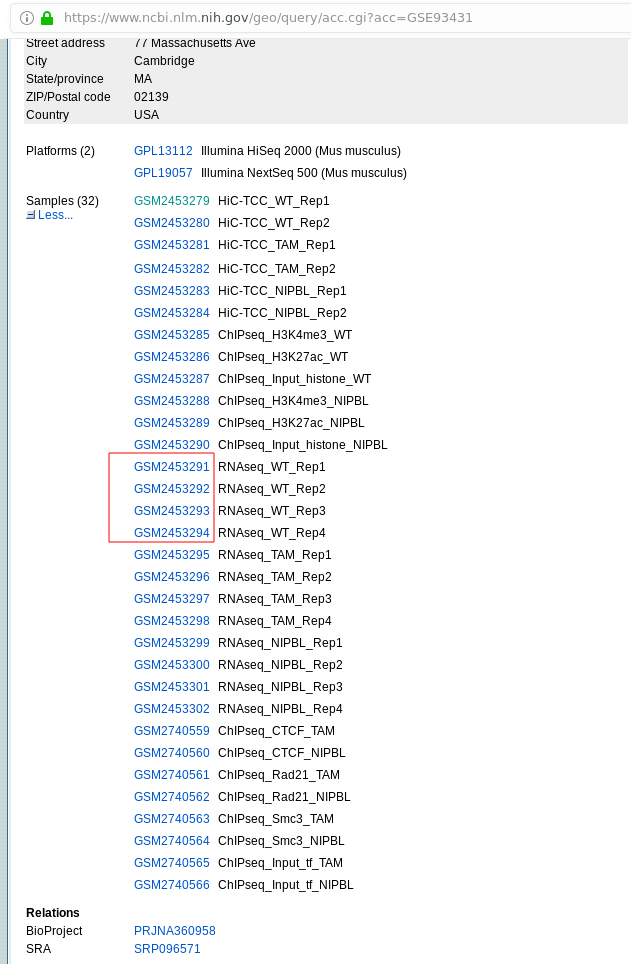
Toi petit étudiant de M1 qui arrive en premier jour de stage… Viens par ici… Oui TOI ! Toi à qui ton maître de stage te demande de récupérer les données de séquençage d'un article vachement bien, sans que tu saches le faire… TOI ! Toi le physicien qui se met à la biologie mais qui ignore…
-
Rendre ses projets R plus accessibles grâce à Shiny
Vous avez un script que vous souhaitez partager avec une équipe expérimentale ? Vous ne voulez pas que les utilisateurs modifient le code pour paramétrer votre programme ? Vous codez avec R ? Alors cet article est fait pour vous ! Nous allons voir comment créer une application web avec R et permettre à votre utilisateur d’exécuter votre code…
-
Du CV jusqu'au poster avec Inkscape (débutant)
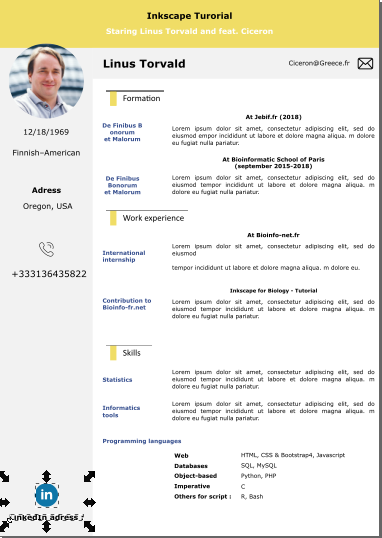
Nous revoilà pour de nouvelles aventures sur Inkscape ! Le but de ce tuto est moins de faire son CV avec Inkscape, quand des outils qu’on utilise tous les jours le font très bien, que de se familiariser avec un outil puissant en manipulant des notions de bases qui peuvent servir ensuite notamment dans l’élaboration de…
-
LaTeX : les lettres (de motivation) !

Après avoir appris à compiler, à insérer des flottants, à mettre en forme les paragraphes et à insérer des mathématiques, il est temps de faire une petite pause hors de la classe article et de se pencher sur les lettres (notamment parce que si mes prédictions sont justes, certain·e·s d'entre vous doivent écrire des lettres…
-
Inkscape pour biologistes
Fini les rectangles pour faire des protéines ou les images pixelisées chopées sur des sites douteux ! Dans 10 minutes vous serez un pro d'Inkscape qui est un logiciel gratuit pour "dessiner" avec une prise en main très rapide permettant de réaliser des figures vectorielles, non construites en pixels, pouvant être alors redimensionnées à l'infini en conservant…

