Étiquette : Ensembl
-
R : Convertir des Ensembl IDs en symboles de gènes

Lorsque l'on traite des données de RNA-seq, il arrive très souvent de se retrouver avec une matrice de quantification de l'expression des gènes (un tableau avec le nombre de reads par gène) dont le nom des gènes est représenté par leur identifiant (ou ID) de chez Ensembl (ex. : "ENSG00000128573") et non par leurs symboles…
-
L'analyse de données RNA-seq : mode d'emploi

Un jour, un biologiste se pointe chez vous avec d'une part un disque dur externe dans la main, d'autre part l'air soucieux. Il veut que vous analysiez ses données RNA-seq. Le disque, c'est parce qu'il a environ 50Gb de données à vous transmettre ; l'air soucieux, c'est parce qu'elles ont coûté dans les 15'000 euros, et…
-
À la découverte de BioMart !

Dans le cadre de mon travail, j'ai récemment découvert un outil formidable pour la consultation et la récupération de données à partir de certaines banques : BioMart. Après avoir passé la frustration de devoir utiliser l'interface du service fourni pour télécharger les différentes données dont j'avais besoin, je me suis renseignée davantage sur ce logiciel. De…
-
Débuter avec l'API Ensembl (Perl)
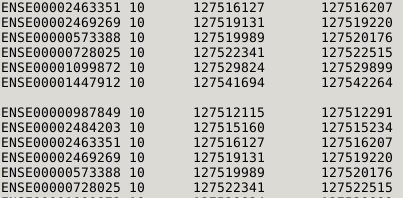
But : Découvrir comment on peut accéder très simplement aux informations d'Ensembl à l'aide d'un script Perl. Niveau : débutant. Pré-requis : Avoir des notions de Perl allant jusqu'à l'utilisation de librairies. Avoir accès à une machine où les librairies Perl d'Ensembl sont installées. Parmi les sites agrégeant de l'information biologique, Ensembl fournit une API (Application Programming Interface)…

