Étiquette : PCA
-
Représenter rapidement une ACP avec R et ggplot2
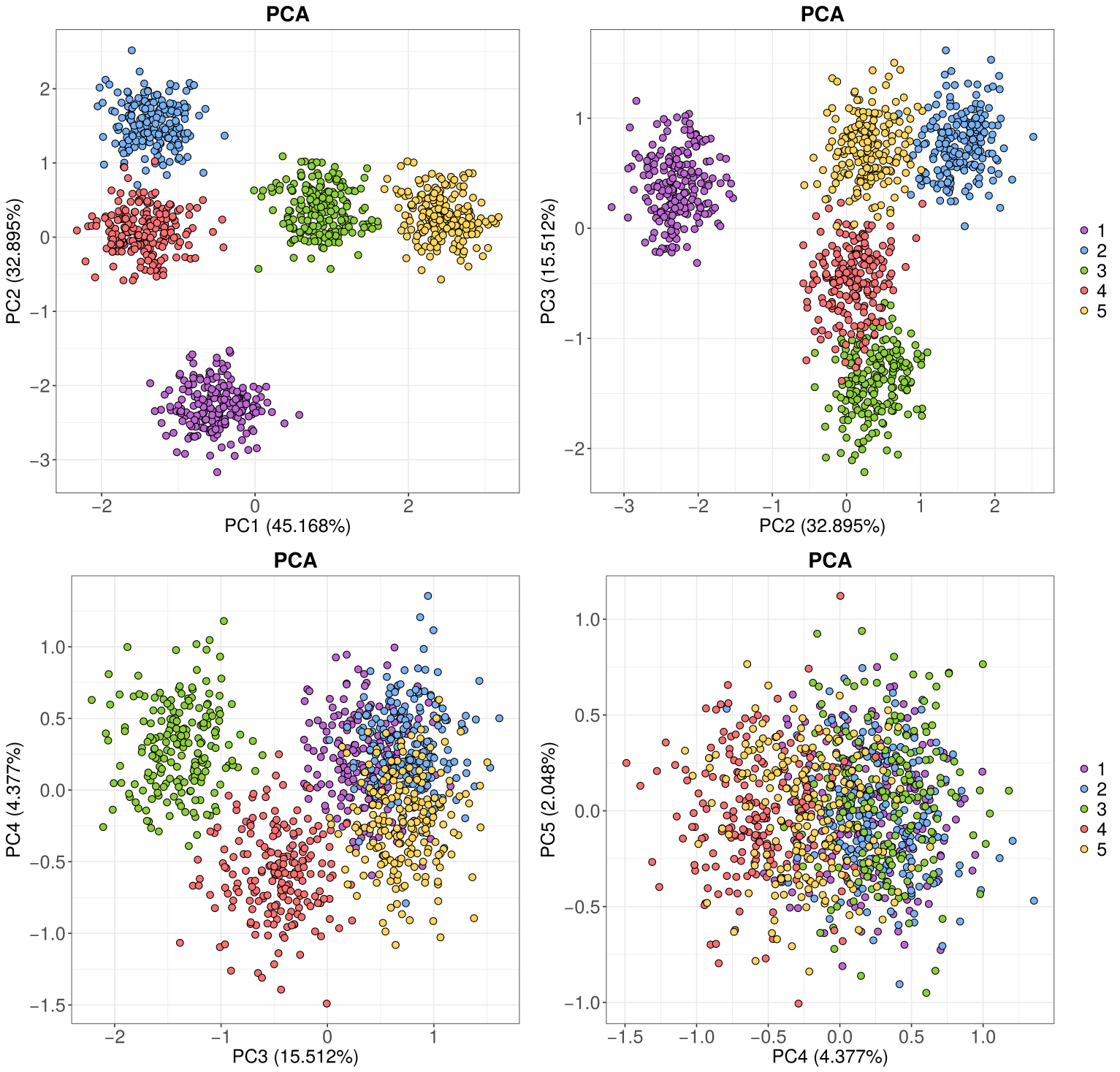
Je ne sais pas pour vous, mais moi, à chaque fois que j'assiste à une réunion de labo, il y a quasi systématiquement un graphique d'ACP pour montrer les données. Et à chaque fois, il s'agit d'un graphique de base, généré avec R, avec la fonction plot(), des couleurs qui piquent les yeux et des…
-
La bière décodée dans un Hackuarium

Vous aimez la bière et la science ? Vous voulez connaître la composition de votre mousse favorite ? Vous avez envie de goûter de nouvelles bières proches de celles que vous connaissez, ou complètement différentes ? Il y a quelques temps déjà, je vous ai décrit la place de la bioinformatique dans les laboratoires citoyens. Aujourd'hui, un projet sympathique…
-
L'analyse en composantes principales (avec R)

L'ACP, ou Analyse en Composantes Principales, est une méthode d'exploration de données qui consiste à réduire la dimensionnalité du problème pour en extraire l'essentiel. Par une projection dans un espace plus petit, on réduit le nombre de variables, et si on réduit suffisamment on peut en faire un outil de diagnostic graphique. Comme c'est une…

