Étiquette : RNA-seq
-
R : Convertir des Ensembl IDs en symboles de gènes

Lorsque l'on traite des données de RNA-seq, il arrive très souvent de se retrouver avec une matrice de quantification de l'expression des gènes (un tableau avec le nombre de reads par gène) dont le nom des gènes est représenté par leur identifiant (ou ID) de chez Ensembl (ex. : "ENSG00000128573") et non par leurs symboles…
-
La transcriptomique spatiale
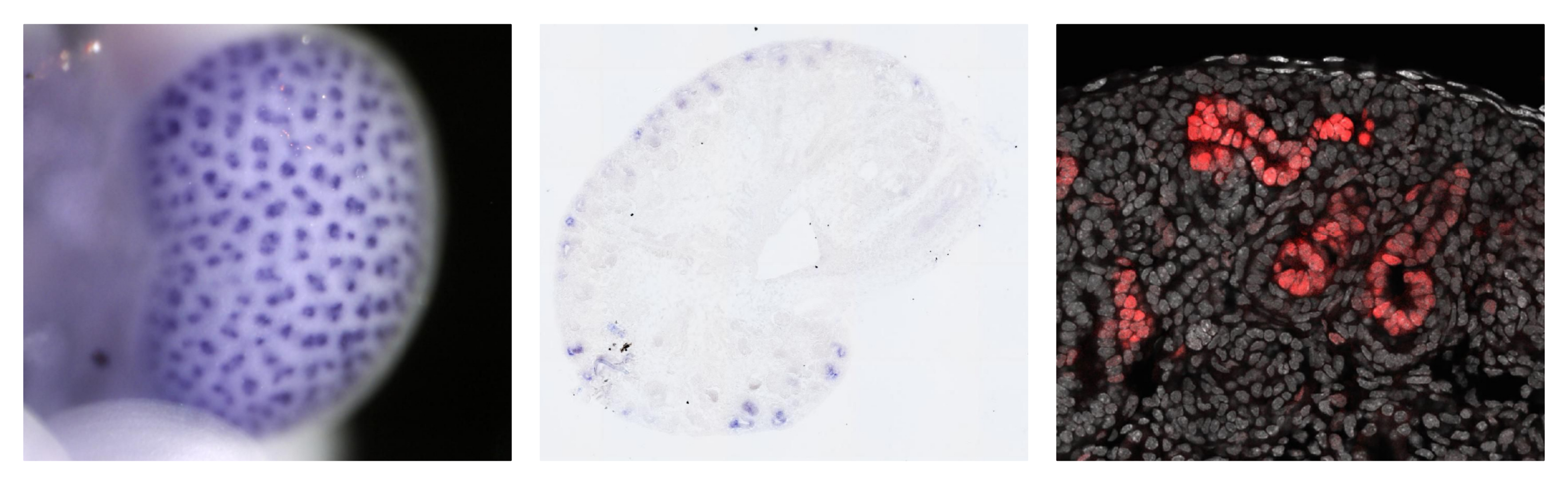
Non, on ne va pas partir faire du RNA-seq dans la station spatiale internationale, rassurez-vous. Je vais vous parler de cette (relativement) nouvelle technique qui permet en une seule expérience de mesurer l'expression des gènes et de localiser cette expression dans un organe plus ou moins complexe. Pour faire une analyse à large échelle du…
-
L'annotation de régions génomiques et les analyses d’enrichissement

Les annotations sont essentielles lors d'analyses fonctionnelles à large échelle sur le génome. Lorsque l’on pratique des analyses en génomique, basées sur des techniques comme le RNA-seq ou le ChIP-seq, on se retrouve avec respectivement une liste de transcrits ou de pics (régions génomiques). Dans le cas des analyses de ChIP-seq, on souhaite caractériser les gènes cibles du…
-
RNA-seq : plus de profondeur ou plus d'échantillons ?

Lorsque l'on se lance dans l'aventure du séquençage haut débit de transcriptome, on est amené à se poser LA question, oui LA, celle que l'on redoute à peu près tous quand on a un budget serré : À quelle profondeur dois-je séquencer mes échantillons ? Toutes les publications s'accordent à le dire, plus on a de réplicats,…
-
L'analyse de données RNA-seq : mode d'emploi

Un jour, un biologiste se pointe chez vous avec d'une part un disque dur externe dans la main, d'autre part l'air soucieux. Il veut que vous analysiez ses données RNA-seq. Le disque, c'est parce qu'il a environ 50Gb de données à vous transmettre ; l'air soucieux, c'est parce qu'elles ont coûté dans les 15'000 euros, et…
-
Analyse des données de séquençage à ARN
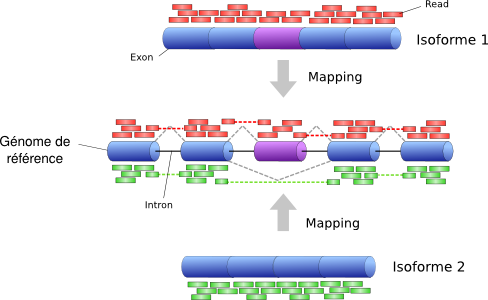
Depuis quelques années, le domaine du séquençage de l'information génétique est rentré dans une nouvelle ère : “le séquençage de seconde génération”. Cette avancée technologique a permis une analyse plus en profondeur de l'ADN et l'ARN. Nous pouvons citer parmi ces nouvelles technologies le ChIP-seq (Chromatine Immuno Precipitation sequencing) ou RNA-seq (Séquençage à ARN). Le projet…

