Durant mon stage de M2, j’ai eu l’occasion de chatouiller ce drôle d’animal qu’est pandas. En effet, j’ai travaillé sur des données de protéomique contenues dans des fichiers tabulés. Il s'agissait de comparer la présence des protéines ou leur expression dans différents échantillons. Les abondances relatives (la variable étudiée) étaient indiquées pour les différentes protéines identifiées (plusieurs milliers et correspondant aux lignes du fichier) dans les différents échantillons analysés (correspondant aux colonnes). J’ai donc dû manipuler ces données tabulées pour en tirer des conclusions vis-à-vis du comportement des protéines selon les différents échantillons. Je viens ainsi vous présenter ce qu’est pandas et quelques commandes pour pouvoir l’utiliser « de façon basique » et assez facilement afin de commencer rapidement à analyser ses données. Comme vous l'aurez compris, il s’agira d’un article introductif.
Mais qu’est-ce que pandas ?
Non, ce n’est pas vraiment un ursidé qui m’a aidée à trifouiller dans les données mises à ma disposition. Ce n’est pas non plus des « Troubles neuropsychiatriques pédiatriques auto-immunes associés à une infection à streptocoque » (merci Wikipédia pour la découverte !). Alors qu’est-ce réellement ?

Pandas est une bibliothèque open source qui vise à l’analyse et la manipulation de données en Python. L'objectif est de permettre la manipulation de grands volumes de données structurées (tabulées, multi-dimensionnelles, voire hétérogènes) et de données temporelles de façon simple mais efficace et rapide dans le but de leur exploitation. La bibliothèque contient donc un grand nombre d'outils pour récupérer, agréger, combiner, transformer, trier, découper, déplacer et exporter les données, ainsi que gérer les valeurs manquantes, calculer des statistiques et produire des graphiques.
Enfin, pandas a été conçu à partir de Numpy et reprend une structure de données utilisée dans R (les data.frame). Il est donc naturel de retrouver des similarités avec ce dernier dans l'utilisation de pandas.
Je tiens à préciser que, hélas, pandas ne sait pas encore faire le café… (Désolée Nolwenn :p) Peut-être durant une prochaine version ! D’ailleurs, vous pouvez très bien proposer des améliorations et participer au développement ici. Il y a même un Gitter et une mailing list ainsi que le traditionnel Stack Overflow pour ceux qui auraient des questions !
Documentation
La documentation officielle est assez imposante avec 2215 pages pour la version PDF mais peut être assez interactive via le site. De plus, je conseille les pages suivantes qui permettent de se plonger dans pandas rapidement pour une utilisation simple sans avoir à digérer la documentation en entier (heureusement pour nous !) :
- Une page qui prétend permettre d’appréhender pandas en 10 minutes
- Des exemples rapides d’utilisation de pandas façon « livre de cuisine »
- De nombreux tutoriels pour les nouveaux utilisateurs ou sur des points précis
Et en bonus, il existe des cheat sheets ! Des fiches qui réunissent les commandes principales et essentielles de pandas. Elles sont très pratiques, à imprimer et afficher à côté de son ordi pour y jeter un coup d’œil rapide et ne pas perdre du temps à chercher la commande sur internet ou dans la doc ! Je vous renvoie vers celle éditée par l'équipe de développement de pandas ici. Plusieurs autres peuvent également être trouvées sur internet (DuckDuckGo est ton ami !).
Installation
À la date de fin d’écriture de l’article, la dernière version (release) de pandas est la 0.22.0 (Février 2018). Elle est supportée par les versions de Python 2.7, 3.5 et 3.6, vous avez donc l’embarras du choix. Avant tout, pour pouvoir « jouer » avec pandas, il faut passer par l’installation. Heureusement pour nous, cette dernière est assez simple puisqu’il s’agit d’un package Python.
Avec Pip (la méthode recommandée avec l'utilisation de virtualenv)
|
1 2 |
#Version 2.X de Python<br> pip install pandas |
#Version 3.X de Python
pip3 install pandas
Avec Conda
On peut également passer par Conda pour l’installation (pour rappel, deux super articles sur cet autre animal : ici et là) :
|
1 |
conda install pandas |
Avec les gestionnaires de paquets
Ou bien en utilisant les gestionnaires de paquets des distributions GNU/Linux en téléchargeant les paquets python-pandas pour Python 2.X et python3-pandas pour Python 3.X. Par exemple, pour une distribution de type Debian ou Ubuntu :
|
1 2 |
# Pour Python 2.X<br> sudo apt-get install python-pandas |
#Pour Python 3.X
sudo apt-get install python3-pandas
Autres façons
Il est évidemment possible de passer par le site officiel. Si vous êtes plus aventurier(ère), vous pouvez également télécharger les codes sources depuis GitHub ou même la source tarball.
En conclusion, pas d’excuse pour ne pas trouver un moyen d’installer pandas qui soit à son goût ! A noter qu'une installation sans virtualenv peut tout de même déstabiliser le sytème d'exploitation (merci Nolwenn !).
Utilisation
Import de la bibliothèque
Avant même de penser à manipuler la bête, il faut l'importer :
|
1 |
import pandas as pd |
Structures de données
Il faut savoir que pandas utilise principalement deux structures de données : la Series (1 dimension) et le DataFrame (2 dimensions). Pour résumer simplement, on va utiliser la structure Series lorsque l'on est en présence de données tabulées présentant plusieurs lignes et une seule colonne de données (de la variable nous intéressant), alors que l'on va plutôt avoir recours au DataFrame à partir de deux colonnes.
Series
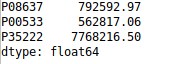
Exemple de fichier de données que l'on peut traiter avec un objet Series :
| Accession | Abondance |
|---|---|
| P08637 | 792592,97 |
| P00533 | 562817,06 |
| P35222 | 7768216,5 |
| … | … |
Au niveau du code :
|
1 |
series = pd.Series(data=[abondance1, abondance2, etc], index=["Prot1", "Prot2", etc]) |
#Par exemple
series = pd.Series(data=[792592.97, 562817.06, 7768216.5], index=["P08637", "P00533", "P35222"])
print(series)
DataFrame
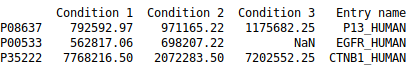
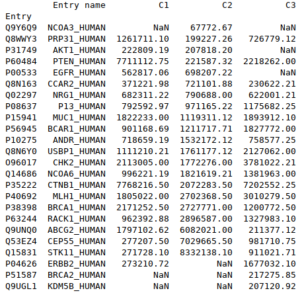
Exemple de fichier de données permettant d'utiliser un DataFrame :
| Accession | Entry name | Condition 1 | Condition 2 | Condition 3 |
|---|---|---|---|---|
| P08637 | P13_HUMAN | 792592,97 | 971165,22 | 1175682,25 |
| P00533 | EGFR_HUMAN | 562817,06 | 698207,22 | |
| P35222 | CTNB1_HUMAN | 7768216,5 | 2072283,5 | 7202552,25 |
| … | … | … | … | … |
Au niveau du code :
|
1 |
df = pd.DataFrame(data, index) |
#Par exemple
idx = ["P08637", "P00533", "P35222"]
donnees = {"Entry name":["P13_HUMAN", "EGFR_HUMAN", "CTNB1_HUMAN"], "Condition 1":[792592.97, 562817.06, 7768216.5], "Condition 2":[971165.22, 698207.22, 2072283.5], "Condition 3":[1175682.25, None, 7202552.25]}
df = pd.DataFrame(data=donnees, index=idx)
print(df)
Concernant le paramètre data, on peut passer des dictionnaires (de Series, arrays, etc), des DataFrames ou des ndarray de numpy (voir la doc).
Différentes Manips avec Pandas
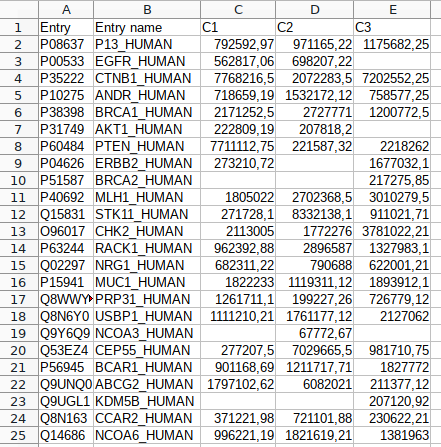
/!\ Ici, je ne traiterai que d'exemples avec des données (à analyser) numériques avec des index non numériques (chaînes de caractères). Je vous invite donc à adapter ces exemples pour des données de nature différente ou hétérogènes (et à compléter cet article avec un autre présentant de nouveaux exemples et nouvelles applications !).
Je travaillerai avec un fichier contenant des données simulées qui se présente comme ceci :
Import des données
À partir d'un fichier EXCEL
Oui, le monde n'est pas (encore) parfait. Il arrive (trop souvent) qu'il faille manipuler des fichiers Excel. Mais rassurez-vous, avec pandas, nous aurons seulement à récupérer les données du fichier et les stocker dans un DataFrame (ou Series s'il n'y a qu'une seule colonne de données). Nous ne toucherons plus à ce type de fichier pour nos traitements.
|
1 2 3 4 5 |
xl = pd.ExcelFile("path/to/file.xlsx")<br> print(xl.sheet_names) # Affiche les noms des différentes feuilles du fichier Excel<br> df = xl.parse("nameOfSheetToAnalyse", index_col=NumberOfTheColumnWhichIsTheIndexColumn)<br> # Ou encore<br> df = xl.parse(sheet_name="nameOfSheetToAnalyse", index_col=NumberOfTheColumnWhichIsTheIndexColumn) |
#Par exemple
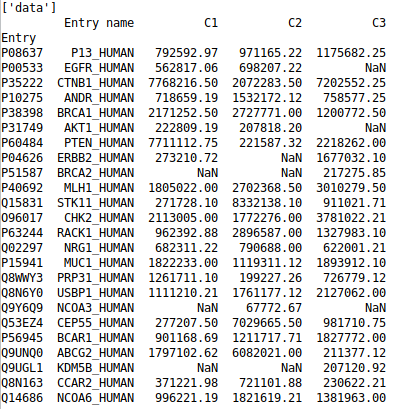
xl = pd.ExcelFile("/media/DATA/Bioinfo-fr/data_excel.xlsx")
print(xl.sheet_names)
df = xl.parse("data", index_col=0)
print(df) # Affiche le dataframe df contenant les données de la feuille 'data' du fichier Excel data_excel.xlsx
Une autre façon de faire est possible en utilisant une seule fonction : read_excel().
|
1 |
df = pd.read_excel("path/to/file.xlsx", sheet_name=NumberOfTheSheetToAnalyseTheDataFrom, index_col=NumberOfTheColumnWhichIsTheIndexColumn) |
#Par exemple
df = pd.read_excel("/media/DATA/Bioinfo-fr/data_excel.xlsx", sheet_name=0, index_col=0)
Apparemment, d'après une réponse sur StackOverflow, la première solution serait plus rapide. On remarque également que l'on peut soit préciser le nom de la feuille à analyser ou directement le numéro de celle-ci, ce qui est utile quand on ne connaît pas les intitulés des feuilles. Cela fonctionne pour les deux solutions, ce n'est pas dépendant de la façon de faire choisie. Je vous laisse aller lire la documentation pour plus de détails.
À partir d'un fichier CSV
Quand on a plus de chance, ce sont des fichiers au format csv que nous avons à notre disposition. De manière analogue à la fonction read_excel(), on utilise la fonction read_csv(). Il y a un paramètre optionnel delimiter='\t' si votre fichier est au format tsv.
|
1 |
df = pd.read_csv("path/to/file.csv", index_col=NumberOfTheColumnWhichIsTheIndexColumn) |
#Par exemple
file_csv = "/media/DATA/Bioinfo-fr/data_CSV.csv"
df = pd.read_csv(file_csv, index_col=0)
Premier aperçu des données
Une fois importées, il est (enfin) possible de jeter un coup d’œil à nos données pour pouvoir commencer à les exploiter. Avec les quelques fonctions ou variables suivantes, nous aurons un rapide aperçu sur ces dernières.
|
1 2 |
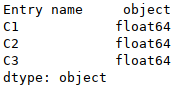
#Renseigne sur le type des données de chaque colonne du dataframe<br> df.dtypes |
|
1 2 3 4 |
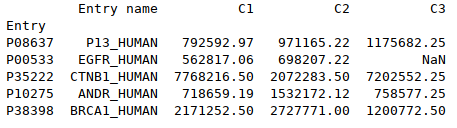
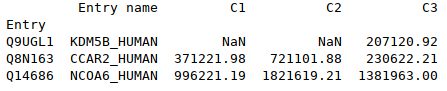
# Sélectionne les premières lignes du dataframe (par défaut 5)<br> df.head()<br> # Sélectionne les dernières lignes du dataframe (par défaut 5)<br> df.tail(3) |
|
1 2 3 4 5 6 |
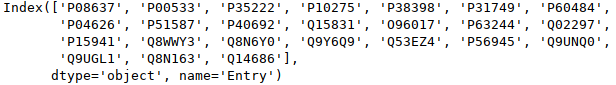
# Sélectionne la colonne des indices du dataframe<br> indices = df.index<br> # Noms des colonnes (hormis celui des indices)<br> colonnes = df.columns<br> # Valeurs des données<br> vals = df.values |

|
1 2 3 |
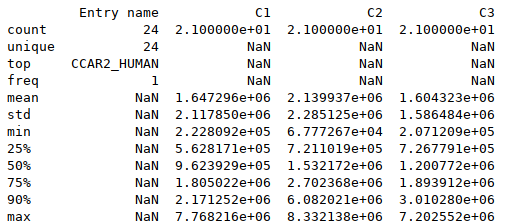
# Renvoie quelques statistiques sur les données sous forme d'un dataframe<br> # percentiles : par défaut 25%, 50% et 75% ; include : colonnes à inclure dans les statistiques (par défaut toutes les colonne de données numériques) ; exclude : colonnes à exclure (par défaut aucune)<br> df.describe(percentiles=[.25, .5, .75, .9], include='all'; exclude=None) |
# Renvoie la moyenne des données (numériques) par colonne
df.mean() # ou df.mean(0)
# moyenne par ligne
df.mean(1)
Traitements basiques
Après avoir eu une vision globale de nos données grâce à quelques statistiques, nous pouvons les traiter à l'aide des informations obtenues précédemment.
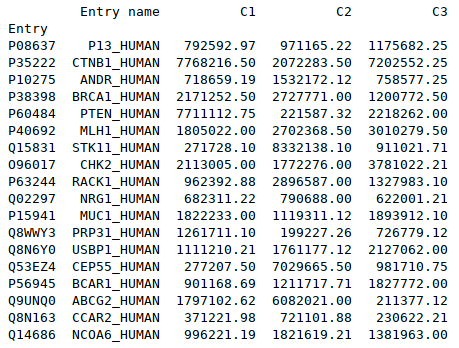
Gérer les données manquantes et/ou nulles
|
1 2 3 4 5 |
# Renvoie un dataframe sans valeur manquante (si le paramètre inplace est à False)<br> # axis : axe selon lequel supprimer les données (0 : ligne , 1 : colonne)<br> # how : any = si au moins une valeur manquante présente, l'ensemble de la ligne/colonne est supprimée ; all = toutes les valeurs d'un ensemble doivent être manquantes pour être supprimées<br> # inplace : supprime les valeurs dans la dataframe au lieu d'en renvoyer un nouveau<br> df_dropna = df.dropna(axis=0, how='any', inplace=False) |
# Par exemple
df_dropna = df.dropna(how='any')
|
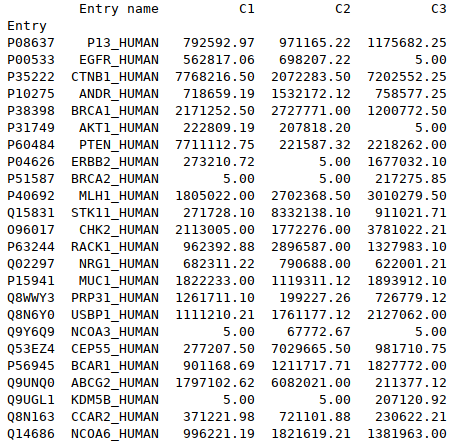
1 2 3 4 |
# Renvoie un dataframe dont les valeurs manquantes ont été remplacées par une valeur précisée<br> df_fillna = df.fillna(value=5)<br> # Renvoie un dataframe avec à la place de chaque valeur un booléen renseignant si la valeur est manquante ou non<br> df_isnull = pd.isnull(df) |
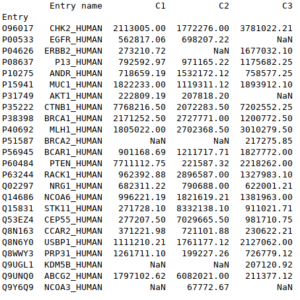
Trier les données
|
1 2 |
# Renvoie un dataframe avec les valeurs triées selon l'axe choisi (indices ou noms colonnes)<br> df.sort_index(axis=0, ascending=True) |
|
1 2 |
# Renvoie le dataframe dont les valeurs sont triées selon les valeurs d'une colonne<br> df.sort_values(by='C2') |
Valorisation des résultats/données
Visualisation
Il est souvent intéressant de pouvoir visualiser ses données sous la forme d'un graphique. Il est possible de créer un graphique directement à partir du dataframe et en utilisant la bibliothèque matplotlib ('import matplotlib.pyplot as plt' en début de script).
|
1 2 3 4 5 |
df.plot()<br> # Affiche le graphique<br> plt.show()<br> # Ou pour sauvegarder<br> plt.savefig("path/to/graph", format="png") |
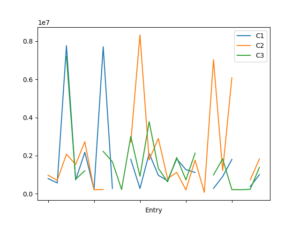
On peut évidemment choisir un type de graphique. Pour cela, il faut utiliser à la place de df.plot() les méthodes associées : df.plot.type() avec 'type' étant 'line', 'bar', 'scatter', 'hist', 'box', etc.
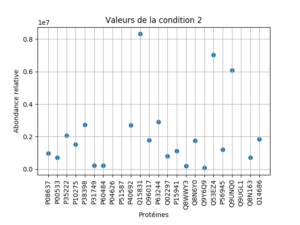
On peut également complexifier un peu selon nos envies et notre maîtrise de matplotlib. Je vous glisse (en cadeau car vous le méritez après avoir lu ce looong article) ici un exemple de graphique dont j'ai eu besoin. J'ai dû modifier la présentation de l'axe des abscisses pour pouvoir visualiser les noms de protéines de façon lisible.
|
1 2 3 4 5 6 7 8 9 10 11 |
N = len(df['C2'])<br> x = np.arange(1, N + 1)<br> width = 1.0<br> plt.scatter(x, df['C2'])<br> plt.xlabel('Protéines')<br> plt.ylabel('Abondance relative')<br> plt.title("Valeurs de la condition 2")<br> plt.xticks(x, df.index, rotation=90)<br> plt.autoscale(tight=False)<br> plt.grid()<br> plt.show() |
Export des résultats/données
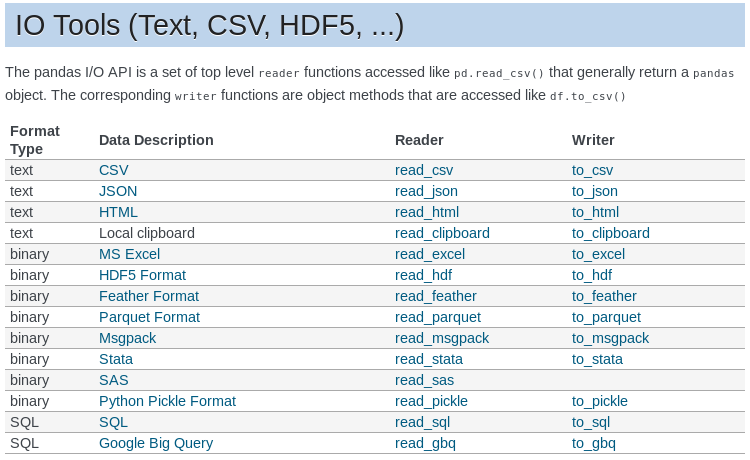
L'export des données est similaire à leur import. Au final, vous pouvez manipuler différents types de fichiers, que ce soit en entrée ou en sortie. Un tableau récapitulatif des possibilités vous permet de le constater (cf documentation).
Informations complémentaires
Il est à noter que pandas peut être utilisé au sein de IPython/notebook Jupiter.
Durant l'écriture de cet article, j'ai même découvert qu'il existait GeoPandas pour manipuler les données géographiques. Donc si vous bossez sur du SIG, pandas est aussi là pour vous ;). Je ne l'ai pas utilisé, mais avec un rapide coup d’œil sur la doc, j’ai pu voir qu'il reposait sur le même principe global avec les types de données Series et DataFrame (GeoSeries et GeoDataFrame).
De plus, pandas offre bien d'autres possibilités et je vous invite à découvrir ces dernières et à venir nous les partager par l'écriture d'un article plus avancé sur l'utilisation de pandas ! Je vous laisse (enfin) vous dépatouiller avec Pandas !
Enfin, je tiens à remercier Olivier Dameron, Nolwenn et lroy qui se sont aventuré(e)s à relire mon article et Chopopope pour sa magnifique aquarelle ! \o/
Références
https://pypi.python.org/pypi/pandas
http://sametmax.com/le-pandas-cest-bon-mangez-en/


Laisser un commentaire