Catégorie : Didacticiel
-
Apprivoiser l'ami ursidé de Python : Pandas

Durant mon stage de M2, j’ai eu l’occasion de chatouiller ce drôle d’animal qu’est pandas. En effet, j’ai travaillé sur des données de protéomique contenues dans des fichiers tabulés. Il s'agissait de comparer la présence des protéines ou leur expression dans différents échantillons. Les abondances relatives (la variable étudiée) étaient indiquées pour les différentes protéines…
-
LaTeX : automatisez le traitement des CSV
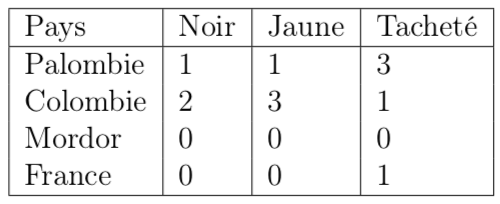
Vous avez peut-être vu l'excellent article de Chopopope sur l'utilisation des flottants en LaTeX, et en particulier la partie sur la création de tableaux. Non ? Pour les retardataires, c'est par ici… Vous vous êtes surement rendu compte que créer des tableaux peut être long et fastidieux. De plus, en bons bioinformaticiens, vos données tabulées sont…
-
Maîtrisez le cache de Rmarkdown !

Pour des raisons de reproduction de la science, il est important de conserver une trace de tout ce que l'on fait sur son ordinateur. Pour cela, faire des rapports est la meilleure manière que je connaisse qui permette d'inclure le code et les résultats d'une analyse. Pour faire ça bien avec R, on a déjà…
-
LaTeX : les maths !

Après avoir appris à compiler, à insérer des flottants et à mettre en forme les paragraphes, on va s'attaquer à un truc plutôt coolish qui fait la force de LaTeX : la mise en forme de maths ! Comme c'est long et vaste, on va faire ça en plusieurs fois 😉 ! Tables des matières : 1. Ce qu'il…
-
Jouer avec l'API de KEGG

Il n'est pas rare que nous ayons un jour besoin de récupérer des informations de la base de données KEGG (Kyoto Encyclopedia of Genes and Genomes). Cette base de données fournit un nombre conséquent d'informations sur les génomes et les réseaux de gènes mais également sur les voies métaboliques ou les maladies. Dans ces cas…
-
Petite introduction sur… les éléments répétés

Suite à l'excellent billet proposé par un autre auteur du blog, de nombreuses questions sur ce que sont les éléments répétés sont restées en suspens. Après le séquençage du génome humain dans les années 2000, de nombreux chercheurs ont constaté que la majeure partie du génome n'était pas composée de gènes, mais d'ADN à l'époque…
-
LaTeX : la mise en forme du texte et des paragraphes
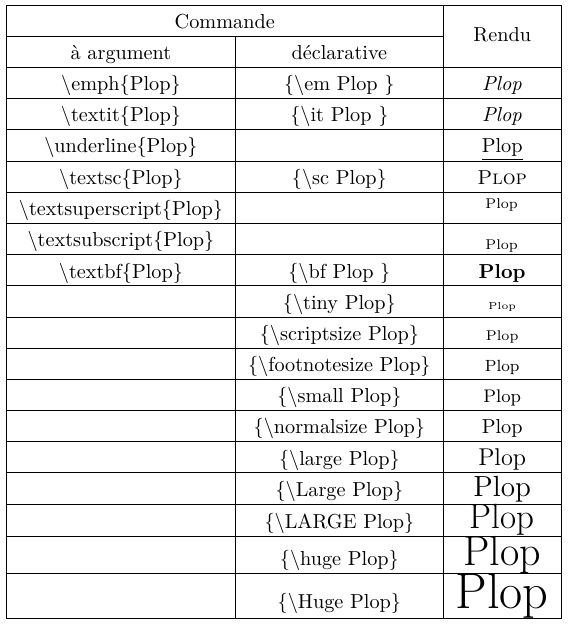
Bon maintenant qu'on sait compiler et insérer des flottants, un truc critique à voir c'est comment mettre en forme : mettre en italique, en emphase, en gras, souligner, faire des listes à puces, insérer du code… Préparez-vous, ça risque d'être un peu long :). (Et par un peu long je veux aussi dire un peu enquiquinant,…
-
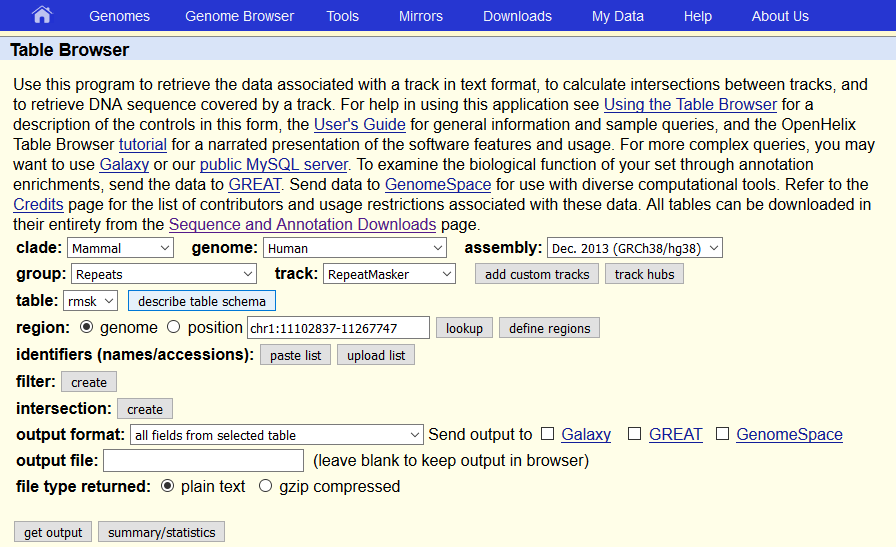
Les éléments répétés du génome humain : aperçu rapide avec R et le tidyverse
Dans un précédent article, nous avions regardé le fichier d'annotation des gènes du génome humain d’après Gencode. J'avais utilisé pour cela la puissante combinaison dplyr + ggplot2 (packages centraux du tidyverse), particulièrement adaptée à tout ce qui est manipulation et visualisation de données tabulaires. Mais notre génome n'est pas constitué que de gènes, loin s'en…

