Étiquette : ADN
-
Manipulation d'intervalles génomiques dans R
Introduction Nous avons abordé, dans le précédent article de cette série, les bases de la manipulation d'intervalles dans R. Ce deuxième article a pour objectif de montrer comment manipuler des intervalles génomiques. Au niveau le plus basique, un intervalle est défini par deux nombres entiers positifs délimitant son début et sa fin. Nous allons pouvoir…
-
Meet-4EU+, le bilan !

Bien le bonjour amis lecteurs. Pour le billet du jour je vous propose une visite dans les coulisses d'un cours/projet déroulé cette année à l'université de la Sorbonne : Meet-4EU+. J'apporterai ainsi mon regard sur l'organisation de cette année, agrémentée de quelques petites anecdotes subjectives. L'idée de ce cours est de proposer aux étudiants de master…
-
1… 2… 3… 4C ! Ou comment capturer l'état de la chromatine.
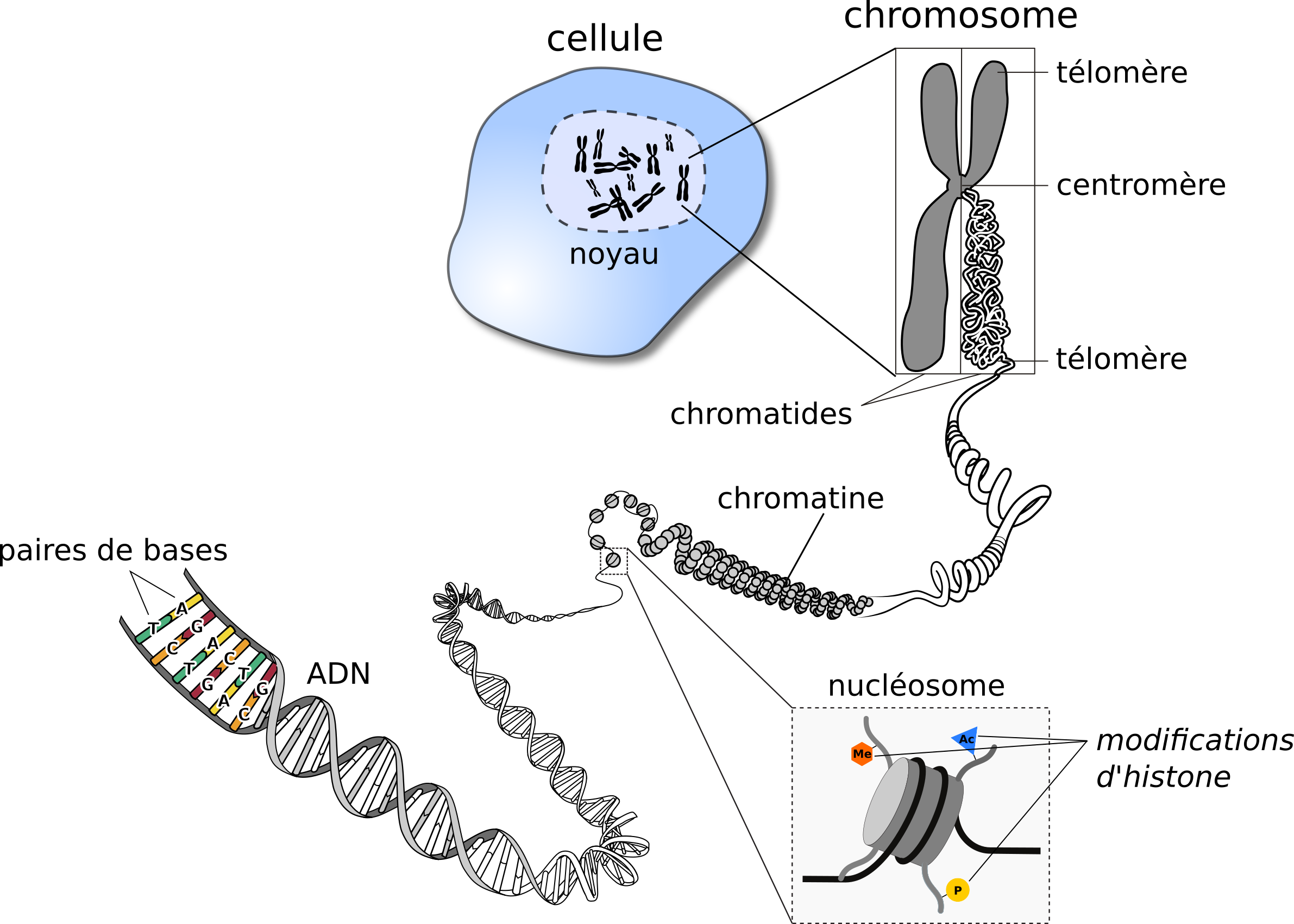
Une cellule eucaryote comporte un noyau qui contient l’information génétique portée par les chromosomes, eux même composés d’ADN. Chez l'Homme, l'ADN des chromosomes mis bouts à bouts mesure 1,9 mètre de long. Or, tout ce matériel génétique doit tenir dans le noyau des cellules, qui lui mesure 5 à 7 micromètres de diamètre (en moyenne…
-
Questions à… Jean-Marc Victor

Retranscription de la Table Ouverte en bioinformatique (TOBi) du mois d'avril 2017 avec Jean-Marc Victor, directeur de recherche CNRS au LPTMC et responsable du GdR ADN. Pouvez-vous vous présenter ?Jean-Marc Victor : Je ne suis pas bioinformaticien. Je suis un peu bio, mais pas trop informaticien. Et je suis physicien. Et il parait que c'est la première fois…
-
Eh toi ! rev_comp, tu l'écris comment ?

Écrire un algorithme de rev_comp ou complément inverse, on l'a tous déjà fait. Aujourd'hui c'est devenu un classique des cours d'algorithmique en étude de bioinformatique, c'est un algorithme simple mais qui demande de savoir utiliser les structures de contrôle de base. À la fois un bon exercice pratique et pédagogique, doit-il cependant rester implémenté comme…
-
Comment détecter, compiler et annoter des facteurs de transcription ?

Après la réunion de rentrée de tous les contributeurs du blog, nous avons décidé d'étendre la rubrique "Journal Club". Ainsi, il est désormais non seulement possible mais aussi fortement recommandé de proposer des billets discutant d'un article en particulier. Nous inaugurons cette extension avec un sujet passionnant : la détection de facteurs de transcription. Bonne lecture…
-
Les éléments transposables : de l’appellation "ADN poubelle" à leur étude.

Les éléments transposables sont des séquences d'ADN mobiles dont l'existence à été mise en évidence par Barbara McClintock dans les années 40 chez le maïs. Ces séquences sont présentes dans toutes les branches de l'arbre du vivant et peuvent représenter une grande partie du génome (environ 40% du génome chez l'Homme et jusqu'à 90% chez…


