Catégorie : Astuce
-
Fabriquer un trackhub dans UCSC

J'ai décidé de partager avec vous la petite astuce du moment que j'ai découverte grâce à Jonathan et que j'ai incorporée dans mon travail actuel (merci encore à lui, il a lu toute l’infâme documentation de UCSC). Le navigateur génomique (pour ne pas dire genome browser) de UCSC nous autorise donc à générer et visualiser…
-
Un bookmarklet pour accéder plus facilement aux publications
Si vous êtes au CNRS, ou à l'Inserm, vous êtes probablement familiers avec BiblioInserm ou BiblioVIE, les portails d’informations et d’accès aux revues de ces institutions. Le portail BiblioInserm Le portail BiblioVIE Ces portails vous permettent d’accéder par exemple à PUBMED et d’y chercher des articles, sous réserve d’avoir un identifiant et le mot…
-
Analyses rapides de fichiers

Langage : shell, sous GNU/Linux Commandes présentées :wc, awk, sed, tr, head, nl, cut Niveau : débutant Dans le cadre de notre travail, nous sommes souvent amenés à manipuler de nombreux fichiers contenant des milliers de lignes et des dizaines de champs. Dans ces cas-là, nous avons souvent tendance à virer à la paranoïa et à vouloir nous…
-
Fusionner des fichiers entre eux : la commande join
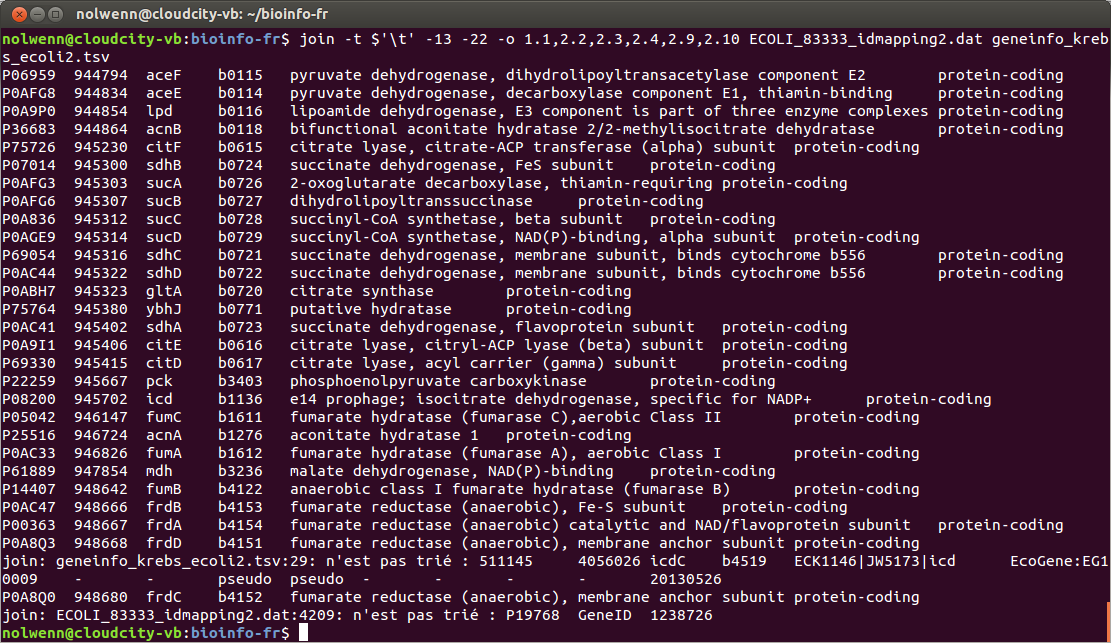
Langage : shell Commande présentée : join Niveau : débutant Présentation de la commande join La commande join est disponible nativement sur les systèmes d'exploitation GNU/Linux. Il s'agit d'une commande POSIX et elle est donc présente sur tous les systèmes d'exploitation UNIX et UNIX-Like. La plupart des gens utilisent cette commande pour récupérer les lignes communes entre deux…
-
SQLite

Dans un précédent article, nous vous avons parlé des bases de données, leur importance et leur intérêt. Ici je vais vous parler de SQLite, une bibliothèque donnant accès à un moteur de base de données relationnelle qui vous permettra de travailler avec du SQL et cela sans avoir besoin de configurer ou d'installer quoi que…
-
Chercher des motifs dans un fichier
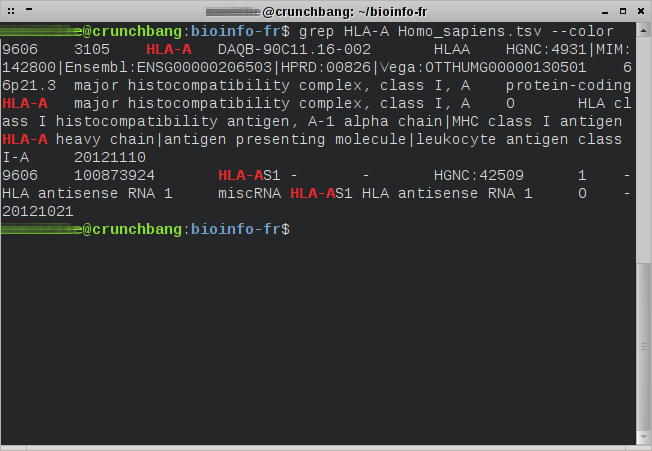
Langage : shellCommandes présentées : grep, split (succintement)Niveau : débutant Présentation de la commande grep La commande grep est disponible nativement sur la plupart des systèmes d'exploitation GNU/Linux. La plupart des utilisateurs utilisent cette commande pour rechercher un mot ou un groupe de mots, que nous appellerons motif (pattern en anglais), dans un fichier texte. Cependant cette commande…
-
Monter un serveur de test pour des besoins d’analyses en bioinformatique

Dans cet article je vais vous parler d'une facette un peu moins connue de la bioinformatique en vous présentant comment il est possible, à l'heure actuelle, d'assembler un serveur d’analyse bioinformatique avec un budget serré. Il ne s'agit pas d'une étude de marché très poussée mais d'un simple exemple du matériel qu'il est possible d'utiliser pour réaliser un serveur de développement performant.…
-
Astuce : ajouter des options dans un script Bash avec getopt
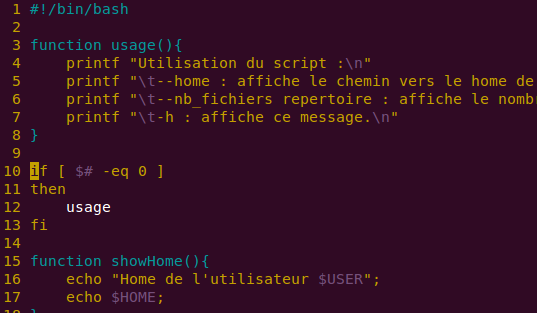
But : comprendre le fonctionnement de getopt en Bash pour éviter la multiplications de script là où un seul générique pourrait suffire. Prérequis : savoir faire des scripts Bash, connaître la substitution de commande et savoir manipuler les arguments. Difficulté : 2 (moyen) Pour ceux qui codent en Perl, vous connaissez déjà sûrement le module GetOpt et plus…

