Catégorie : Astuce
-
Inkscape : l'outil idéal pour vos figures et posters

Je sais pas pour vous mais dans mon labo, le premier réflexe de mes collègues lorsqu'il faut faire une figure ou un poster c'est soit de dégainer la suite Ad*be, soit d'ouvrir P*werPoint. Quel est le problème me direz-vous ? Outre le fait qu'ils soient très coûteux pour le labo/l'université/l'entreprise où vous travaillez, ces outils ne…
-
Créer sa carte géographique avec R

Aujourd’hui je vais vous montrer comment, en utilisant R, on peut faire de belles cartes géographiques. Et là, vous allez me demander, mais pourquoi faire des cartes géographique ? Et pourquoi avec R ? Et bien imaginons que, vous, bioinformaticien de terrain, soyez allé échantillonner des animaux à l’autre bout du monde sur plusieurs sites, par exemple…
-
L'annotation de régions génomiques et les analyses d’enrichissement

Les annotations sont essentielles lors d'analyses fonctionnelles à large échelle sur le génome. Lorsque l’on pratique des analyses en génomique, basées sur des techniques comme le RNA-seq ou le ChIP-seq, on se retrouve avec respectivement une liste de transcrits ou de pics (régions génomiques). Dans le cas des analyses de ChIP-seq, on souhaite caractériser les gènes cibles du…
-
RNA-seq : plus de profondeur ou plus d'échantillons ?

Lorsque l'on se lance dans l'aventure du séquençage haut débit de transcriptome, on est amené à se poser LA question, oui LA, celle que l'on redoute à peu près tous quand on a un budget serré : À quelle profondeur dois-je séquencer mes échantillons ? Toutes les publications s'accordent à le dire, plus on a de réplicats,…
-
Ping en JavaScript (jQuery.ajax)

Dans cet article, nous allons découvrir un moyen de vous faire gagner du temps à travers deux notions étrangères à la Biologie : Le ping et Ajax. Pour les termes techniques, reportez vous au glossaire juste après l'introduction. Mesurer le délai entre un serveur et un client web (on utilisera facilement anglicisme "ping", francisé en "pinguer")…
-
The Bio Code : guide du bon broinformaticien

Malgré la multitude d’outils déjà existants, les occasions d'écrire du code sont nombreuses en bioinformatique. Hormis pour les pousse-boutons avertis, le développement fait souvent partie du quotidien d’un bioinformaticien. Personnellement, c’est une activité qui me plaît beaucoup dans ce métier. Développer ses propres applications et outils apporte toujours une certaine satisfaction (et quand ça fonctionne,…
-
Quelques pistes pour contrôler vos données de ChIP-seq

Le ChIP-seq est une méthode aujourd'hui répandue qui consiste à cibler une partie du génome grâce à une protéine et à séquencer uniquement les parties du génome auxquelles celle-ci s'est fixée. On la capture ensuite avec un anticorps spécifique et on séquence uniquement l'ADN qu'elle protégeait (voir notre article : DNase-seq, FAIRE-seq, ChIP-seq, trois outils d'analyse de…
-
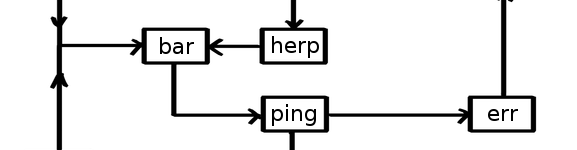
Formaliser ses protocoles avec Snakemake
Vous avez dit protocole ? Qui dit biologie et bioinformatique dit protocole expérimental. C'est le cœur de la démarche scientifique, et un formalisme adapté est la clef pour assurer la reproductibilité des expériences, et ainsi garantir la validation des découvertes par la communauté. En paillasse, les solutions pour formaliser et conserver les protocoles sont plutôt naturellement…

