Catégorie : Découverte
-
1… 2… 3… 4C ! Ou comment capturer l'état de la chromatine.
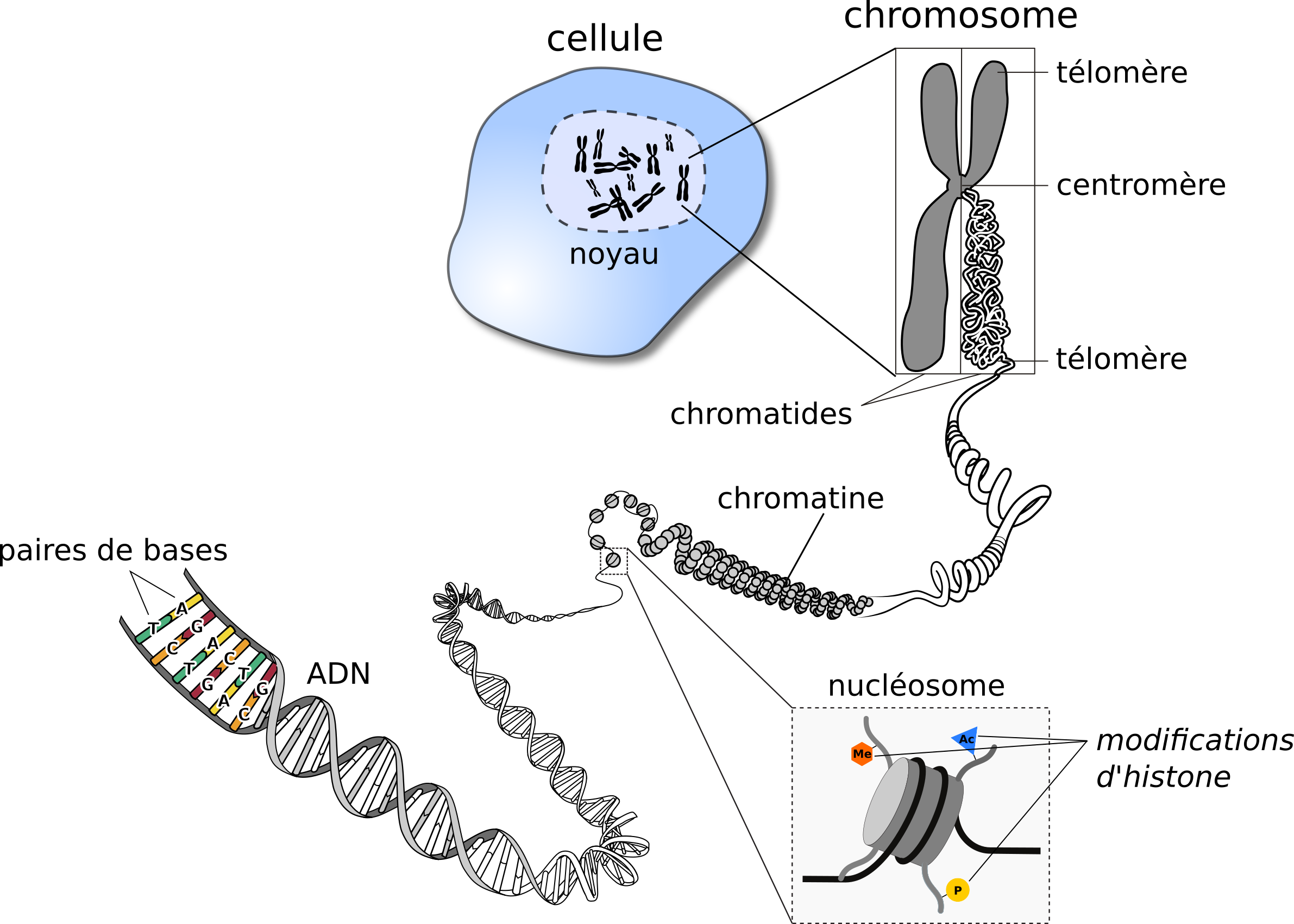
Une cellule eucaryote comporte un noyau qui contient l’information génétique portée par les chromosomes, eux même composés d’ADN. Chez l'Homme, l'ADN des chromosomes mis bouts à bouts mesure 1,9 mètre de long. Or, tout ce matériel génétique doit tenir dans le noyau des cellules, qui lui mesure 5 à 7 micromètres de diamètre (en moyenne…
-
Contrarié par les diagrammes de Venn ? Découvrez les diagrammes UpSet
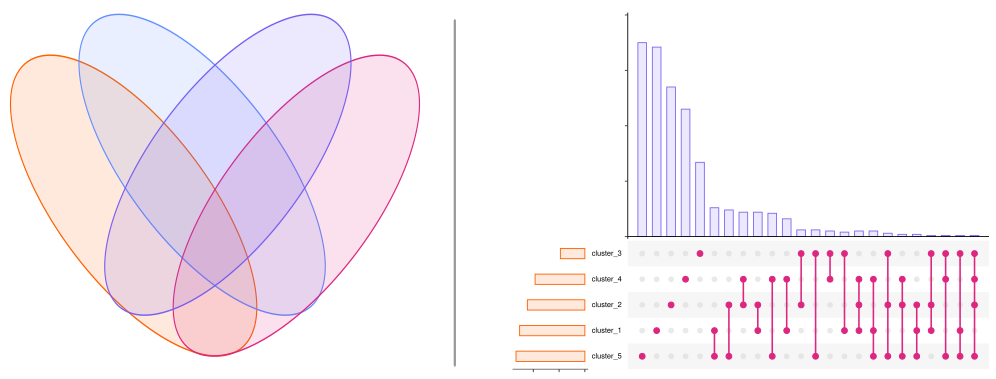
Titre incluant un moyen mnémotechnique ;D Avec ses cercles entrecroisés, on ne présente plus le célèbre diagramme de Venn. Cette représentation est utilisée dans le cas où l'on souhaite représenter le recoupement de données de nombre fini selon plusieurs variables qualitatives. De façon plus simple lorsqu'on a 2 variables qualitatives : combien d'éléments présents dans la…
-
Génomique des paysages

Introduction « Génomique des paysages » cela sonne comme le titre d’une œuvre d’Eduardo Kac. Ce nom un peu post-moderne désigne en fait une discipline scientifique qui a connu une expansion fulgurante au cours de la dernière décennie. Les enjeux environnementaux et de conversation actuels ont rendus pressante la nécessité de mieux comprendre et décrire les espèces…
-
Sept problèmes fascinants posés par les récepteurs olfactifs
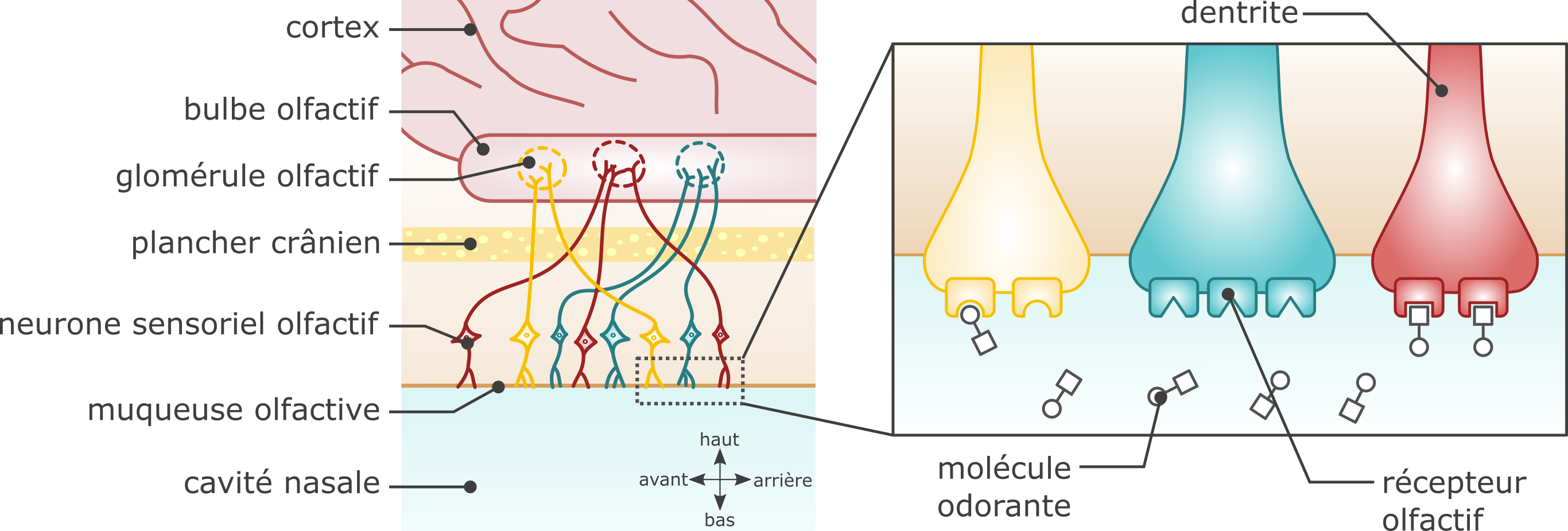
Le cinquième va vous étonner ! Introduction : l'olfaction, un sens assez bien compris et compréhensible L’olfaction n'est peut-être pas le plus noble des sens, comparé à la vue ou l’ouïe par exemple, mais il s'agit d'un sens assez bien compris aujourd'hui. C'est notamment grâce aux travaux des biologistes Linda B. Buck et Richard Axel, récompensés par…
-
Les tests en bioinformatique

Tester est-ce douter ? Aujourd'hui on va parler d'un truc très connu des informaticiens mais encore trop peu connu en bio-informatique : les tests. Cette pratique est pourtant conseillée dans le guide du bon broinformaticien . Alors, qu'est ce qu'un test ? Un test désigne une procédure de vérification d'un système. Son objectif principal est d'identifier un nombre…
-
Rendre ses projets R plus accessibles grâce à Shiny
Vous avez un script que vous souhaitez partager avec une équipe expérimentale ? Vous ne voulez pas que les utilisateurs modifient le code pour paramétrer votre programme ? Vous codez avec R ? Alors cet article est fait pour vous ! Nous allons voir comment créer une application web avec R et permettre à votre utilisateur d’exécuter votre code…
-
La programmation concurrente en python

Ce tutoriel est une traduction infidèle d'un article de realpython.com https://realpython.com/python-concurrency/#when-to-use-concurrency Merci à eux pour leur formidable travail et leur autorisation. Vous avez certainement entendu parler de la librairie asyncio qui a été ajouté à Python 3 et vous êtes curieux de savoir comment elle se place par rapport aux autres méthodes de programmations concurrentes ?…
-
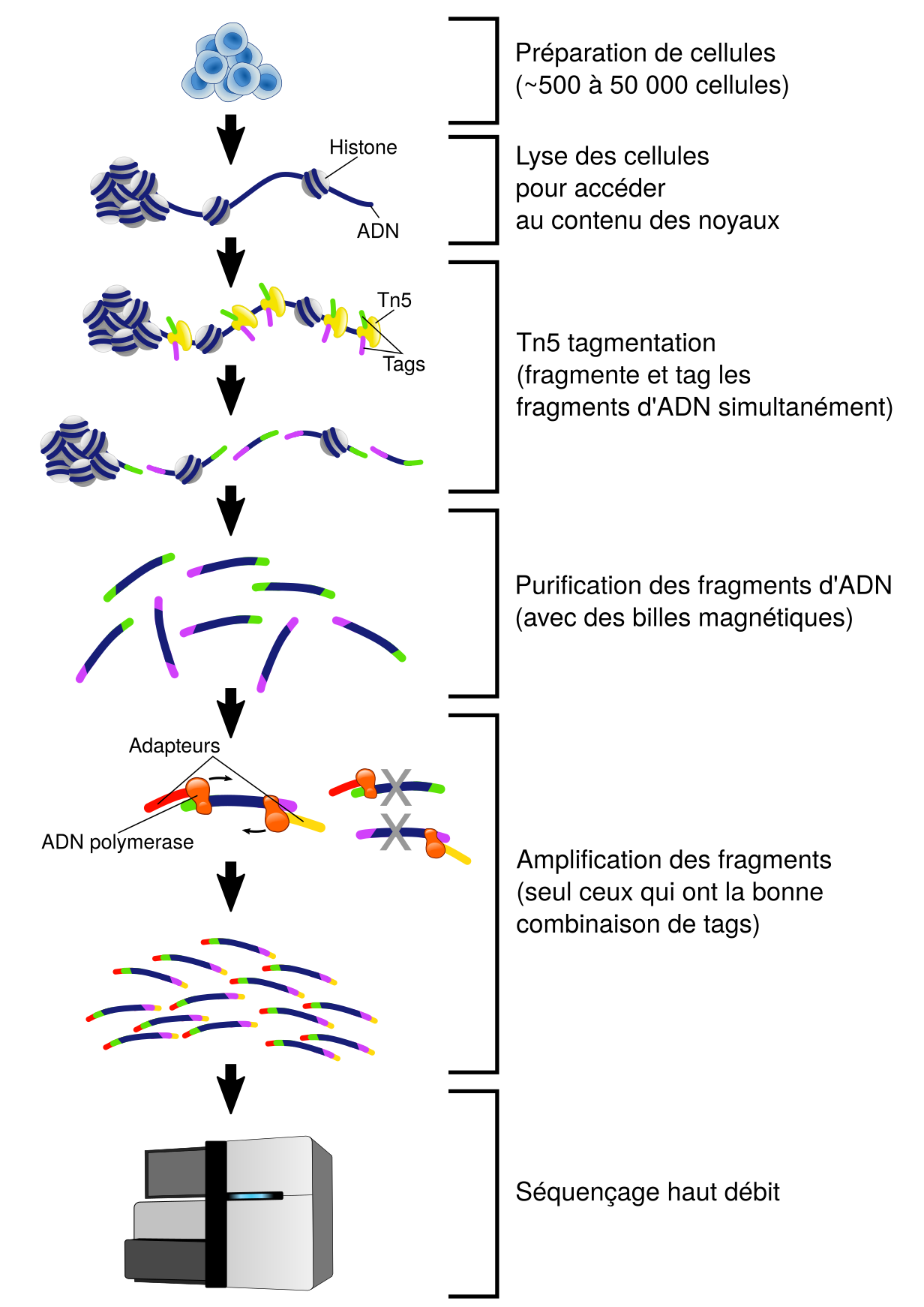
Traquer les régions ouvertes de l'ADN avec l'ATAC-seq
L'étude de la régulation de l'expression des gènes est une discipline complexe qui recoupe des données provenant de divers types d'expériences. Dans un précédent article, nous avions vu trois techniques de biologie moléculaire couplées à du séquençage haut débit classiquement employées pour mettre en évidence les régions accessibles de l'ADN, et donc potentiellement des régions…

