Catégorie : Découverte
-
ViLoVar : un outil pour la visualisation de variations génétiques
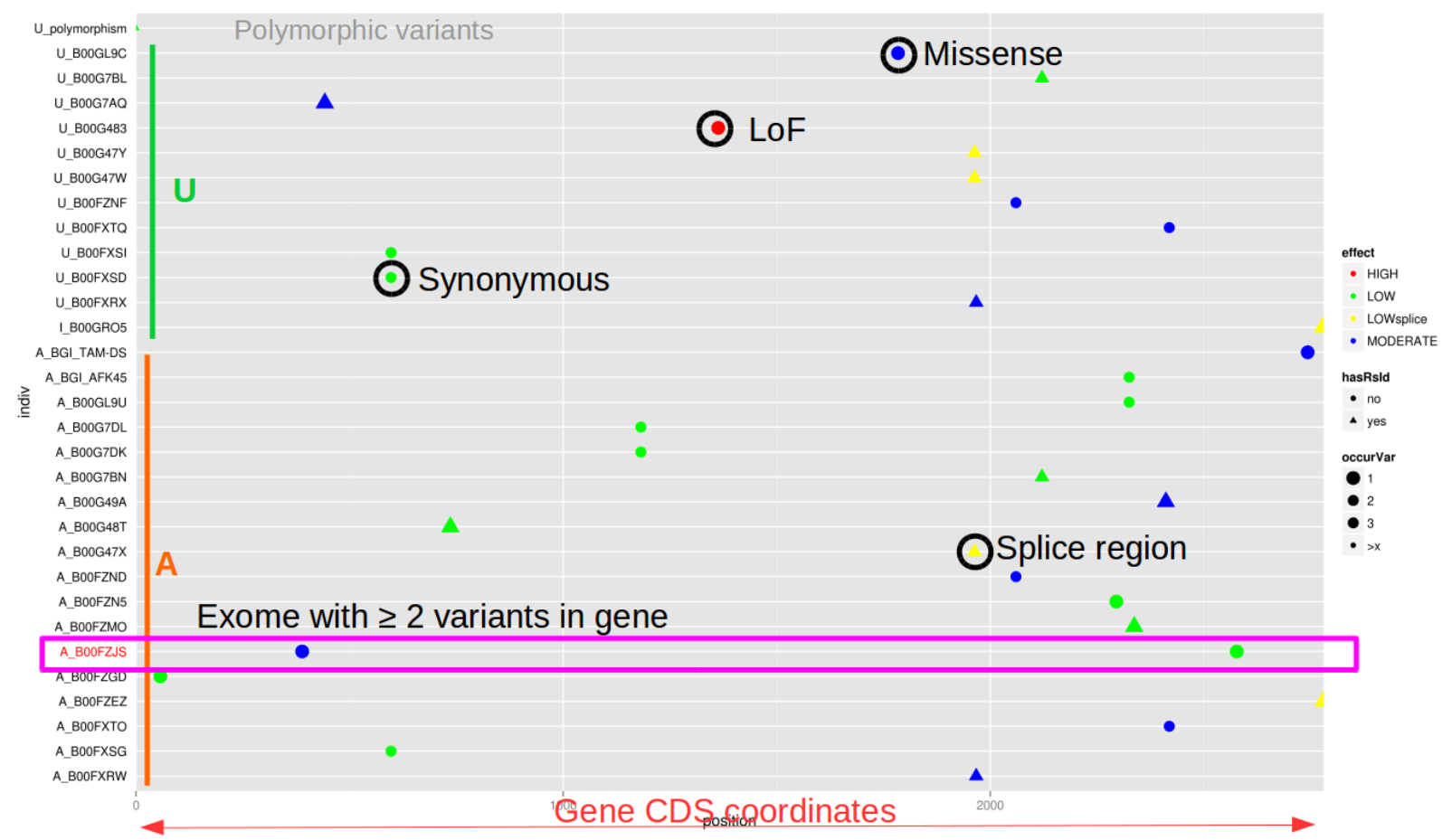
Pour mon premier article, je vais vous présenter un outil que j'ai développé lorsque je travaillais sur le projet "Myocapture"; un projet national de séquençage d'exomes qui portait sur les myopathies (https://www.afm-telethon.fr/myopathie-congenitale-6675). Ce projet visait à trouver de nouvelles mutations responsables de ces maladies rares. Il a également permis d'identifier de nouveaux gènes impliqués dans…
-
Bioconvert - simplifier les conversions de formats
Bioconvert Qui n'a jamais eu à convertir un fichier de données biologiques dans un autre format ? Il y a bien sur le classique fastq vers fasta, pour lequel nombre d'entre nous ont codé un convertisseur "maison", pas forcément optimal. D'autres formats sont parfois plus problématiques, par exemple la conversion vers et depuis GFF2/GFF3. De ces différents…
-
canSnippet : le voilà !

Nous vous l'avions annoncé il y a quasiment un an jour pour jour lors de notre présentation à JOBIM2017 à Lille. Il est maintenant là, disponible, consultable et à portée de tous : canSnippet Community Edition. A vos marques pages, c'est ici que ça se passera dorénavant : https://cansnippet.bioinfo-fr.net/ ! Le principe Avoir une collection de snippets…
-
Apprivoiser l'ami ursidé de Python : Pandas

Durant mon stage de M2, j’ai eu l’occasion de chatouiller ce drôle d’animal qu’est pandas. En effet, j’ai travaillé sur des données de protéomique contenues dans des fichiers tabulés. Il s'agissait de comparer la présence des protéines ou leur expression dans différents échantillons. Les abondances relatives (la variable étudiée) étaient indiquées pour les différentes protéines…
-
Qu'est-ce qu'un site d'initiation de la transcription ?
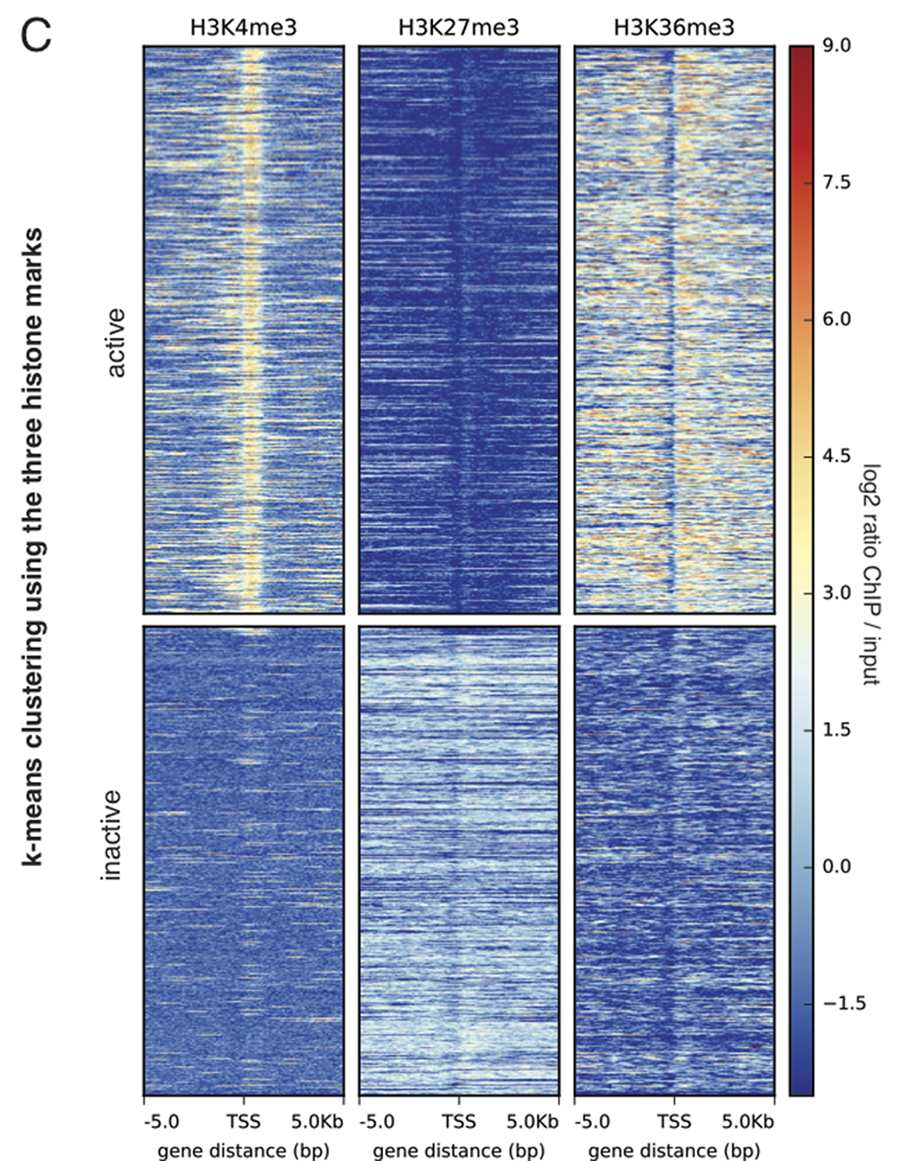
Comment je me suis posé la question. Chez les eucaryotes, l'ADN est organisé en domaines plus ou moins compactés, avec des taux de transcription plus ou moins élevés, et qui sont marqués différentiellement par un certain nombre de marques épigénétiques (méthylation de l'ADN, modifications post-traductionnelles des histones, variants d'histones, etc.). Il est fréquent d'essayer de…
-
Les bases de données de séquençage : GEO, SRA, ENA, ArrayExpress
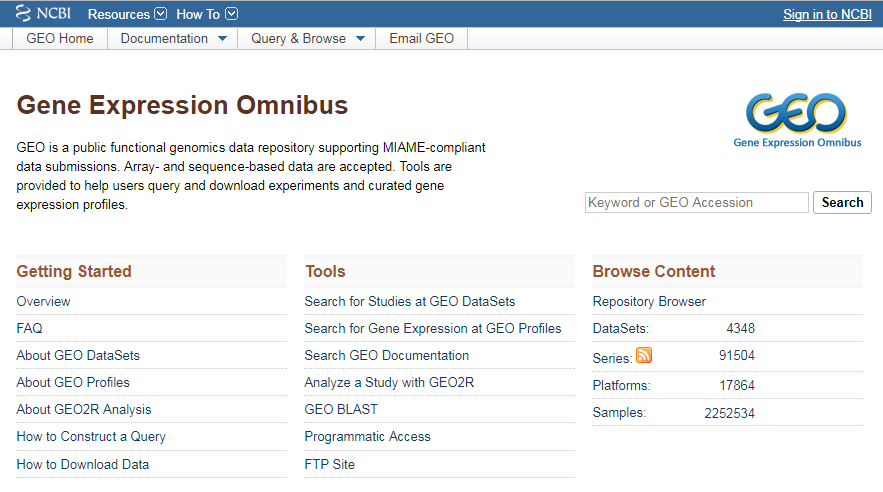
De nos jours, lors de la publication de résultats, il est nécessaire de rendre public les éventuelles données de séquençage générées. Si un faible nombre d’irréductibles continuent à ne fournir les données que sur demande, les bonnes pratiques poussent à les déposer dans des bases de données librement accessibles. Quatre grandes bases de données de séquençage…
-
Le site du NCBI "un peu" plus moderne grâce aux extensions de navigateurs

« Pour la semaine prochaine vous allez devoir trouver et étudier l’article sur le site du NCBI [insérer un nom bien compliqué ici] » Passée la déception d’avoir à se plonger dans un article obscur en anglais, on ouvre l’article en question et là… on découvre un énorme pavé de texte ma foi fort intéressant mais…
-
Créez vos documents collaboratifs en LaTeX

Aujourd'hui, on vous présente une méthode pour créer vos documents collaboratifs en ligne, en utilisant LaTeX, ainsi que quelques astuces qui pourront peut-être vous simplifier la vie ! Un petit peu de contexte Imaginons : vous êtes un jeune chercheur dynamique, et vous voulez rédiger un papier avec vos collaborateurs. Ou bien vous êtes un étudiant, et…

