Étiquette : visualisation
-
R : représenter des genomic tracks avec Gviz
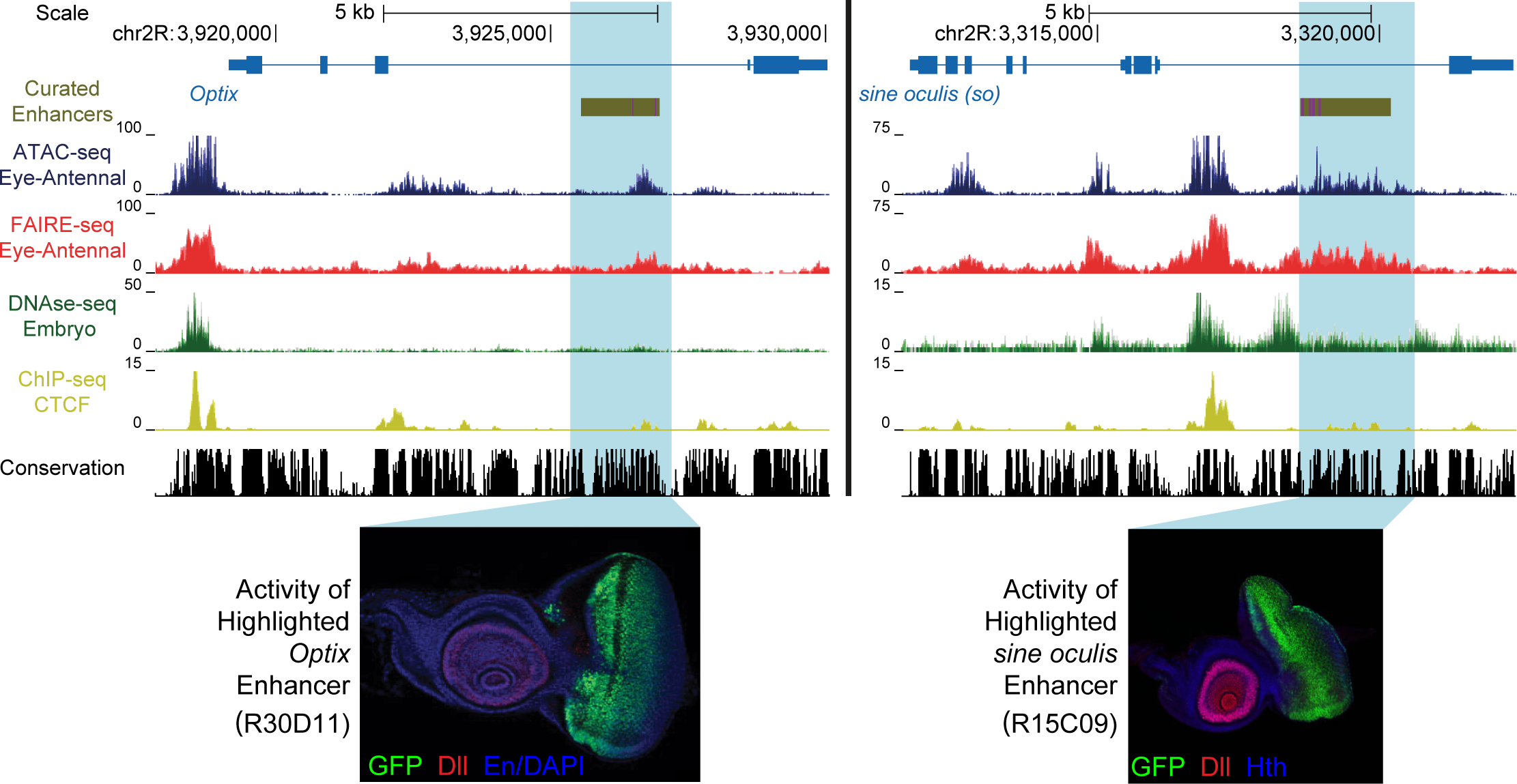
Si vous analysez des données d'épigénomique telles que de l'ATAC-seq ou des ChIP-seq, vous souhaitez sûrement pouvoir représenter des exemples de pics sous forme de genomic tracks comme on en voit souvent dans les publications. Lorsque l'on inspecte ses données de coverage (ou couverture), on charge généralement le fichier Bam ou le fichier BigWig dans…
-
Créer des Heatmaps à partir de grosses matrices en R
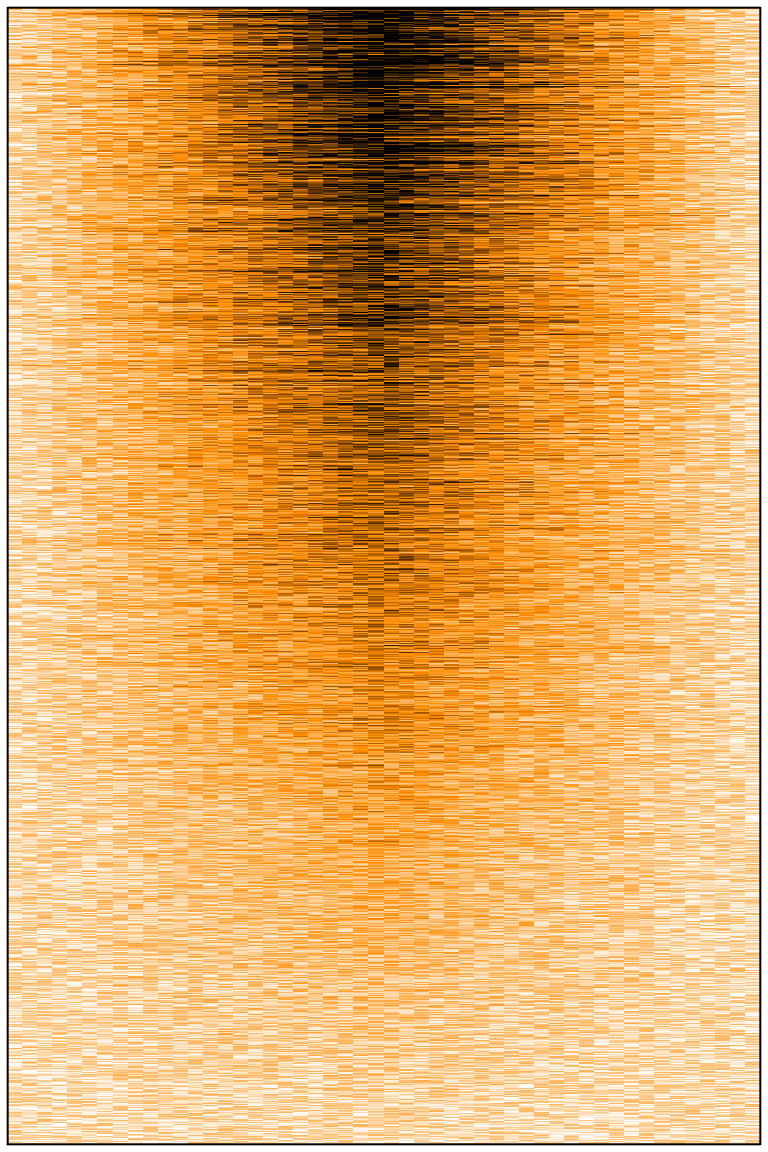
En génomique, et sans doute dans tout un tas d'autres domaines omiques ou big data, nous essayons souvent de tracer des grosses matrices sous forme d'heatmap. Par grosse matrice, j'entends une matrice dont le nombre de lignes et/ou de colonnes est plus grand que le nombre de pixels sur l'écran que vous utilisez. Par exemples,…
-
Contrarié par les diagrammes de Venn ? Découvrez les diagrammes UpSet
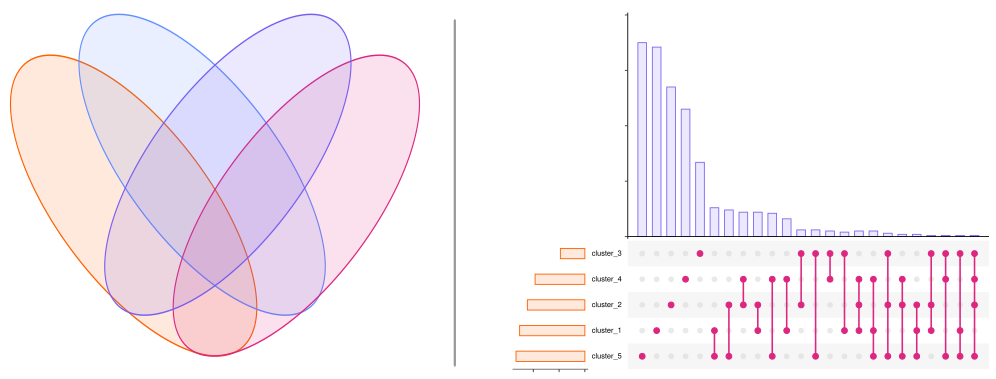
Titre incluant un moyen mnémotechnique ;D Avec ses cercles entrecroisés, on ne présente plus le célèbre diagramme de Venn. Cette représentation est utilisée dans le cas où l'on souhaite représenter le recoupement de données de nombre fini selon plusieurs variables qualitatives. De façon plus simple lorsqu'on a 2 variables qualitatives : combien d'éléments présents dans la…
-
ViLoVar : un outil pour la visualisation de variations génétiques
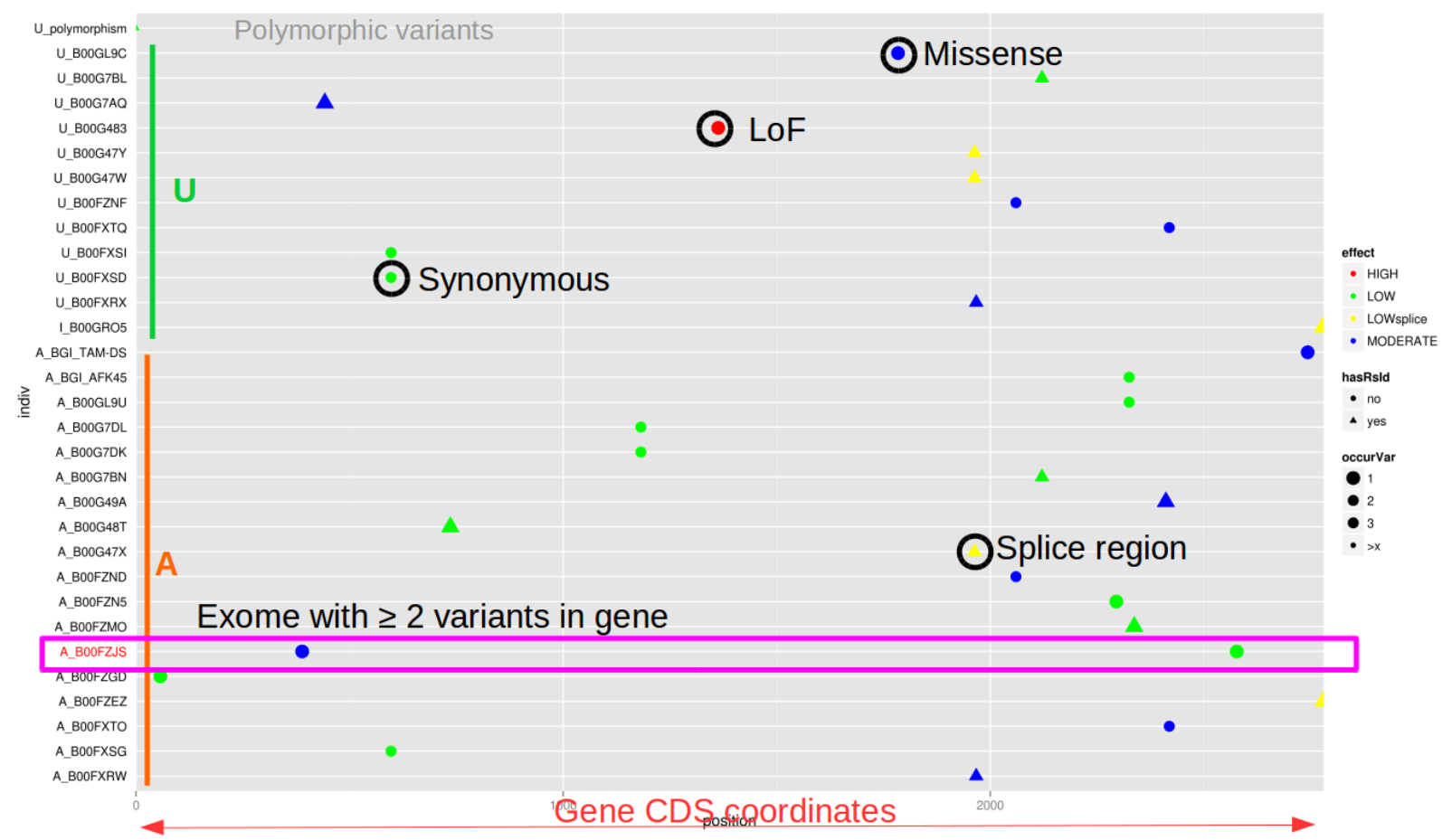
Pour mon premier article, je vais vous présenter un outil que j'ai développé lorsque je travaillais sur le projet "Myocapture"; un projet national de séquençage d'exomes qui portait sur les myopathies (https://www.afm-telethon.fr/myopathie-congenitale-6675). Ce projet visait à trouver de nouvelles mutations responsables de ces maladies rares. Il a également permis d'identifier de nouveaux gènes impliqués dans…
-
Tour d'horizon des outils de visualisation des réseaux biologiques

Après les différentes méthodes d'analyse et de représentation de réseaux métaboliques biologiques, je vais vous parler des différents outils de visualisation. Car oui, la visualisation d'un réseau ou de ses sous-parties peut être le début de son analyse, car elle permet de se rendre compte de sa topologie, de sa complexité, sa connectivité… En biologie, on peut…
-
Gephi pour la visualisation et l'analyse de graphes

Gephi est un logiciel de visualisation et d'analyse de graphes. Il est distribué sous les licences CDDL 1.0 et GPLv3, et est disponible sur les principaux systèmes d'exploitation. Gephi est prévu d'emblée pour tous types de graphes (pas seulement en bioinformatique) dans les principaux formats. Tulip et Cytoscape sont des outils similaires. Cependant, chez moi…
-
Fabriquer un trackhub dans UCSC

J'ai décidé de partager avec vous la petite astuce du moment que j'ai découverte grâce à Jonathan et que j'ai incorporée dans mon travail actuel (merci encore à lui, il a lu toute l’infâme documentation de UCSC). Le navigateur génomique (pour ne pas dire genome browser) de UCSC nous autorise donc à générer et visualiser…