Catégorie : Didacticiel
-
L'annotation de régions génomiques et les analyses d’enrichissement

Les annotations sont essentielles lors d'analyses fonctionnelles à large échelle sur le génome. Lorsque l’on pratique des analyses en génomique, basées sur des techniques comme le RNA-seq ou le ChIP-seq, on se retrouve avec respectivement une liste de transcrits ou de pics (régions génomiques). Dans le cas des analyses de ChIP-seq, on souhaite caractériser les gènes cibles du…
-
SARTools : l'analyse différentielle pour tous
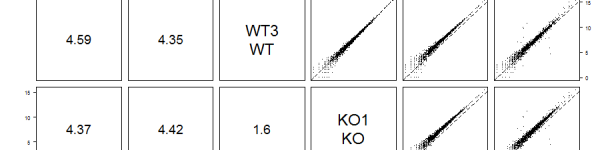
Un article court aujourd’hui pour présenter SARTools, un outil d’analyse différentielle de données de RNA-seq. Plus qu’un outil, SARTools est plutôt un pipeline simplifié pour traiter des données d’analyses réalisées dans un plan d’expérience assez basique. En effet le cadre d’utilisation de SARTools est volontairement limité aux plans d’expérience simples, à savoir des comparaisons de…
-
L'analyse en composantes principales (avec R)

L'ACP, ou Analyse en Composantes Principales, est une méthode d'exploration de données qui consiste à réduire la dimensionnalité du problème pour en extraire l'essentiel. Par une projection dans un espace plus petit, on réduit le nombre de variables, et si on réduit suffisamment on peut en faire un outil de diagnostic graphique. Comme c'est une…
-
Python fait la numba

Suite à cet article, j'ai eu envie de comparer les temps d'exécution de Cython et Numba.Il est toujours intéressant de faire des tests de performances (benchmarks) de temps à autre, pour voir si on ne peut pas améliorer certains de nos algorithmes 🙂 Cython est un très bon moyen pour optimiser vos programmes, mais il…
-
Cython : votre programme Python mais 100x plus vite

Python est un langage extrêmement pratique car il est facile à lire et à écrire, comparé à un langage de "bas niveau" et compilé comme le C. D'un autre côté, à l'exécution il est beaucoup plus lent. C'est un compromis entre les deux qu'offre Cython, permettant d'accélérer votre programme d'un facteur 2 à plus de…
-
Introduction à la ligne de commande

Langage : ShellNiveau : Grand débutant Ce tutoriel n'a originellement pas été écrit ni pour ce blog, ni pour des bioinformaticiens, (ni par moi), mais je pense qu'il a tout à fait sa place ici car il donne les clés pour que n'importe qui puisse se familiariser avec la ligne de commande et sera sûrement très utile…
-
Parser des fichiers HTML en Python

Langage : PythonBibliothèques : bioservices, HTMLParser, re (partiellement)Niveau : débutant-intermédiaire Dans un article précédent, je vous ai présenté le module bioservices en Python. Au cours de mon travail j'ai été amenée à récupérer des informations sur les termes Gene Ontology, et notamment sur les relations entre différents termes. Cependant, les formats de fichiers récupérés sont différents en fonction…
-
Gephi pour la visualisation et l'analyse de graphes

Gephi est un logiciel de visualisation et d'analyse de graphes. Il est distribué sous les licences CDDL 1.0 et GPLv3, et est disponible sur les principaux systèmes d'exploitation. Gephi est prévu d'emblée pour tous types de graphes (pas seulement en bioinformatique) dans les principaux formats. Tulip et Cytoscape sont des outils similaires. Cependant, chez moi…

