Étiquette : modélisation
-
Introduction à la structure secondaire des ARN
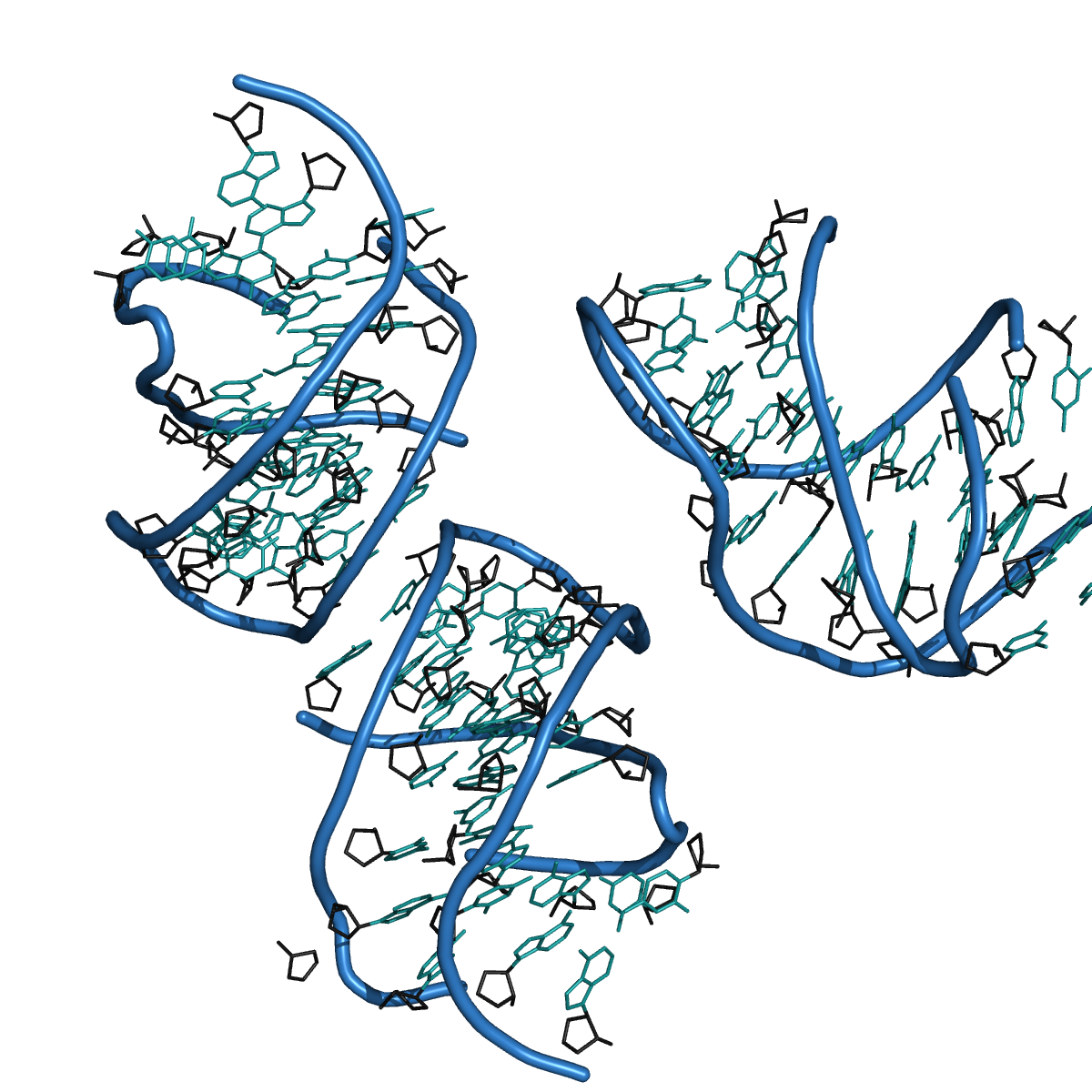
Quand j'étais étudiante en master, la grande majorité des cours de structure étaient réservés aux protéines, ce qui fait que la structure des ARN n'a pas été très développée au cours de mon cursus. Du coup, je vous livre ici une partie de ce que j'ai appris en stage. Définition 0 : ARN L'ARN (acide ribonucléique) est…
-
Champs de force, énergie potentielle et autres joyeusetés en modélisation moléculaire

Introduction Cet article, d'une très longue série, a pour but de donner plus de détails et peut-être - qui sait - vous titiller suffisamment pour enjamber la barrière physique (au sens littéral) et rejoindre le fabuleux monde d'Oz de la bioinformatique structurale. Avant de commencer, je vous conseille de prendre du papier, un crayon, un…
-
Présentation de la licence BISM (ex-MIV) à Lyon 1

[Article publié le 12 mars 2014 et mis à jour le 2 décembre 2015] Présentation La licence BISM (Bio-Informatique, Statistique et Modélisation), anciennement appelée MIV, est un parcours de spécialisation de la licence de Biologie, à l'université Claude Bernard Lyon 1. L’accent de la formation est mis à la fois sur la bioinformatique et sur l'analyse…
-
Introduction aux systèmes multi-agents

Dans le monde merveilleux de l'intelligence artificielle, il existe une catégorie de systèmes de simulation inspirés de la nature et particulièrement adaptés à de nombreuses problématiques en biologie : les systèmes multi-agents. Ces systèmes sont composés d'entités autonomes (les agents) qui interagissent entre elles et avec un environnement dans lequel elles évoluent. Bien évidemment, l'inspiration "biologique"…
-
GGMM 2013 : le bilan

Votre super reporter s'est rendu à deux congrès au mois de Mai, que vous avez pu suivre en direct via des live-tweets. Pour ceux qui ont raté le coche et pour les autre aussi, voici un résumé de ce qui s'est passé durant le premier congrès. GGMM, pour Groupe de Graphisme et Modélisation Moléculaire, est un congrès…
-
La modélisation moléculaire

La modélisation moléculaire est un ensemble de méthodes permettant d'expliquer comment fonctionne le vivant. En effet, le vivant est une succession d’interactions entre différentes molécules : protéines, ADN, ARN, membranes, etc. Ces molécules interagissent les unes par rapport aux autres en fonction de plusieurs paramètres : leurs formes, leurs propriétés chimiques et leur environnement. La modélisation moléculaire permet de prédire la…


