Étiquette : R
-
Rendre ses projets R plus accessibles grâce à Shiny
Vous avez un script que vous souhaitez partager avec une équipe expérimentale ? Vous ne voulez pas que les utilisateurs modifient le code pour paramétrer votre programme ? Vous codez avec R ? Alors cet article est fait pour vous ! Nous allons voir comment créer une application web avec R et permettre à votre utilisateur d’exécuter votre code…
-
Qu'est-ce qu'un site d'initiation de la transcription ?
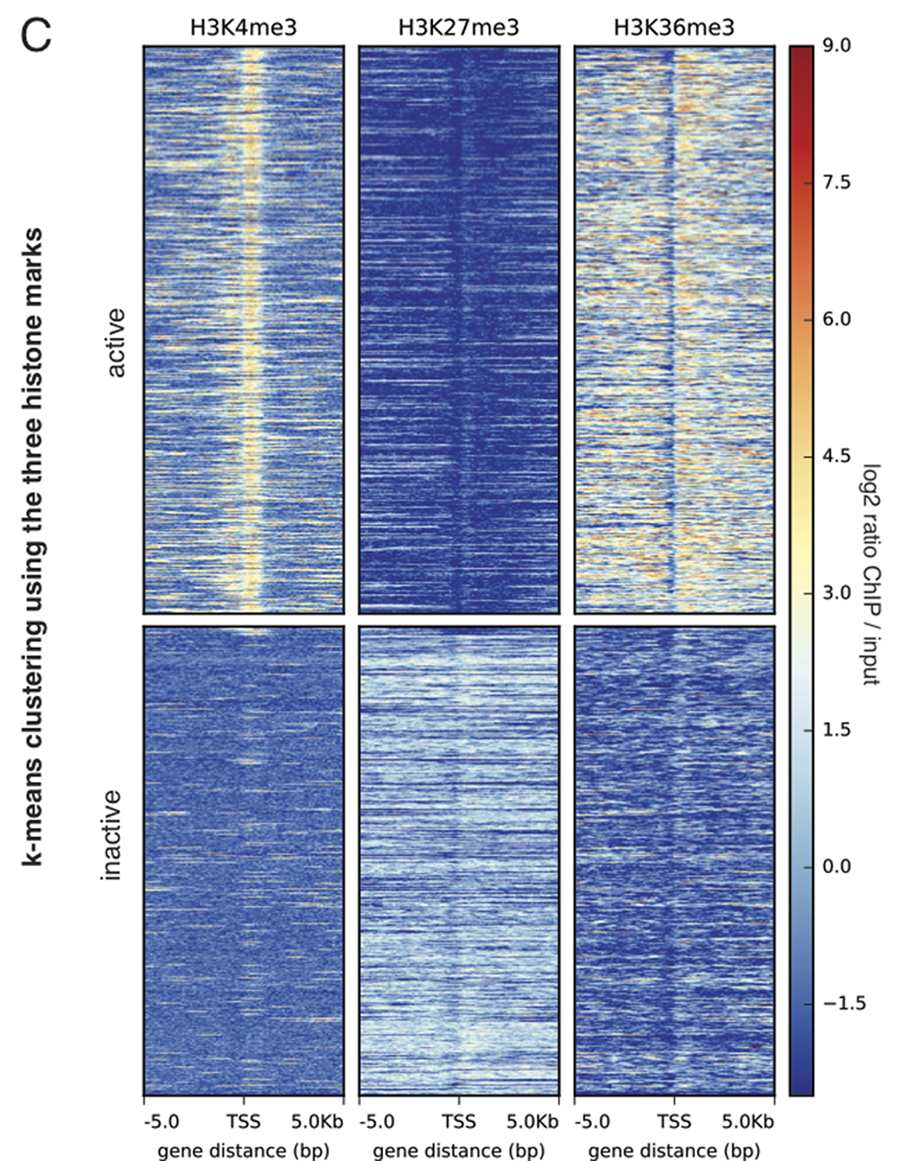
Comment je me suis posé la question. Chez les eucaryotes, l'ADN est organisé en domaines plus ou moins compactés, avec des taux de transcription plus ou moins élevés, et qui sont marqués différentiellement par un certain nombre de marques épigénétiques (méthylation de l'ADN, modifications post-traductionnelles des histones, variants d'histones, etc.). Il est fréquent d'essayer de…
-
Maîtrisez le cache de Rmarkdown !

Pour des raisons de reproduction de la science, il est important de conserver une trace de tout ce que l'on fait sur son ordinateur. Pour cela, faire des rapports est la meilleure manière que je connaisse qui permette d'inclure le code et les résultats d'une analyse. Pour faire ça bien avec R, on a déjà…
-
Les éléments répétés du génome humain : aperçu rapide avec R et le tidyverse
Dans un précédent article, nous avions regardé le fichier d'annotation des gènes du génome humain d’après Gencode. J'avais utilisé pour cela la puissante combinaison dplyr + ggplot2 (packages centraux du tidyverse), particulièrement adaptée à tout ce qui est manipulation et visualisation de données tabulaires. Mais notre génome n'est pas constitué que de gènes, loin s'en…
-
Représenter rapidement une ACP avec R et ggplot2
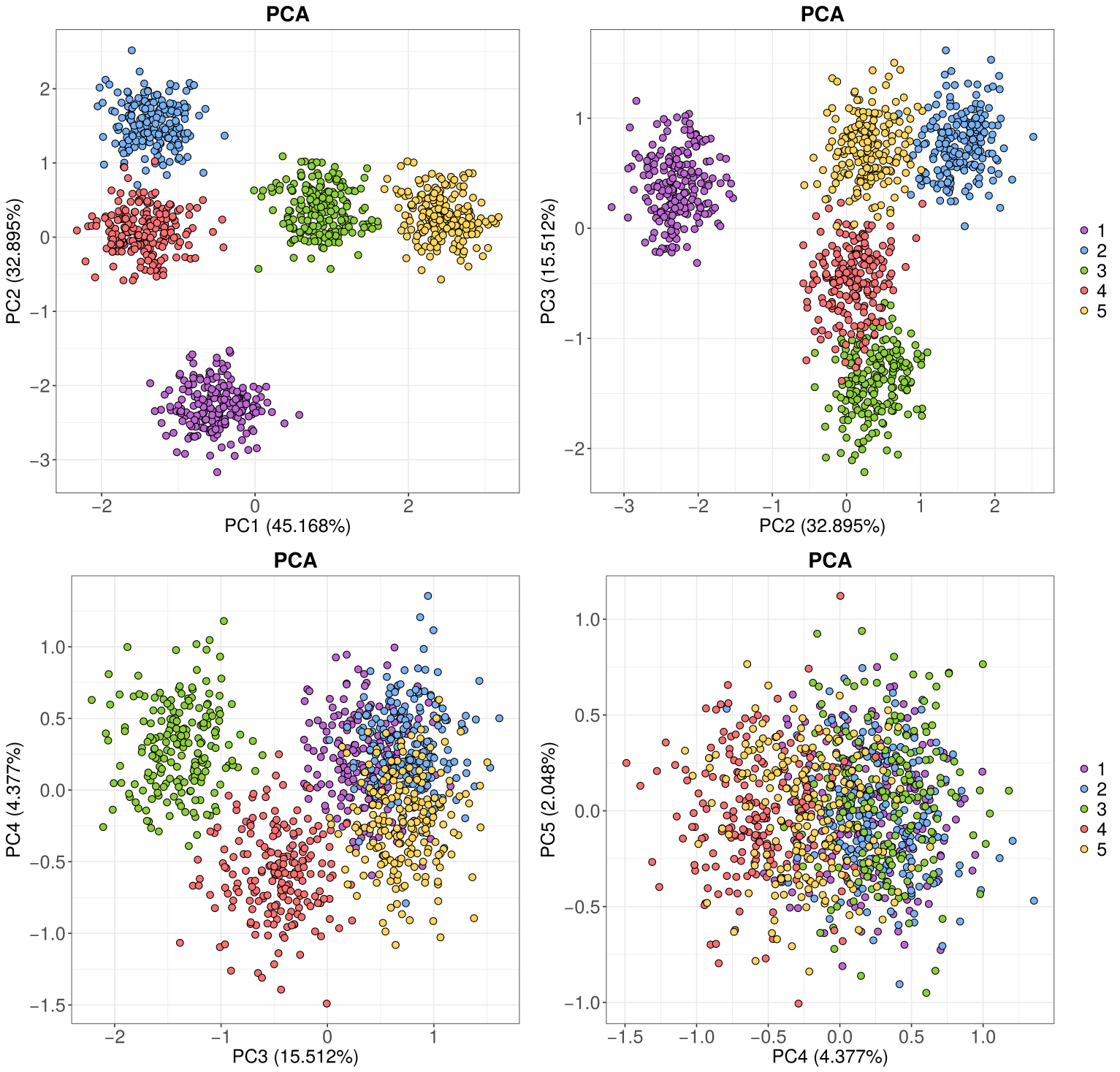
Je ne sais pas pour vous, mais moi, à chaque fois que j'assiste à une réunion de labo, il y a quasi systématiquement un graphique d'ACP pour montrer les données. Et à chaque fois, il s'agit d'un graphique de base, généré avec R, avec la fonction plot(), des couleurs qui piquent les yeux et des…
-
Packrat ou comment gérer ses packages R par projet

Je vais m'arrêter là, je pense que vous avez compris que la gestion de packages sous R est une source d'erreurs faciles. Mais pas d'inquiétude : Packrat fait tout ça , Packrat est simple, Packrat vous veut du bien ! Packrat ? Kézako ? Packrat c'est un petit package R. "Encore un ?!" vous allez me dire, oui mais il…
-
dplyr et le génome humain
Introduction Non, ne fuyez pas tout de suite, chers lecteurs, tout va s'éclaircir : dplyr, c’est plyr pour les data.frame (les tableaux de données). Attendez, j’y viens, plyr, c’est un package R pour appliquer (apply) des fonctions. Donc, dplyr (prononcez “diplir”), c’est un package R, pour appliquer des fonctions à un tableau de données. Et ça,…
-
C'est l'enfeR.

Certains bio-informaticiens ne jurent que par R (j'en fais partie). Je suis amoureux de sa simplicité (sic), son élégance (re-sic), sa documentation et ses innombrables packages tous plus utiles les uns que les autres. Et surtout c'est le seul langage que je maîtrise un peu convenablement, alors forcément je trouve tous les autres langages nuls,…

