Catégorie : Découverte
-
Virtualisez pour plus de reproductibilité

Virtualisez pour plus de reproductibilité Pour commencer Vous avez entendu parler de reproductibilité. Vous voulez vous y mettre ? Vous vous dites que la virtualisation vous aiderait à utiliser toujours la même version d'un outil précis. En savoir plus sur Docker vous aiderait bien ? Cet article est donc pour vous. Je profite de l'occasion pour parler…
-
Identification des relations entre la variabilité génétique et le phénotype : cartographie QTL

Les individus d’une même espèce, à moins qu’ils ne soient des clones identiques, sont tous légèrement différents les uns des autres. Cette différence s’exprime à tous les niveaux, de l’apparence (phénotype macroscopique), au génome (différents allèles pour le même gène), en passant par les phénotypes microscopiques (aussi appelés moléculaires - on pensera ici aux transcriptomes,…
-
Eh toi ! rev_comp, tu l'écris comment ?

Écrire un algorithme de rev_comp ou complément inverse, on l'a tous déjà fait. Aujourd'hui c'est devenu un classique des cours d'algorithmique en étude de bioinformatique, c'est un algorithme simple mais qui demande de savoir utiliser les structures de contrôle de base. À la fois un bon exercice pratique et pédagogique, doit-il cependant rester implémenté comme…
-
Retour d'expérience : bonnes pratiques à appliquer en cas de déréférencement Google (et autres)
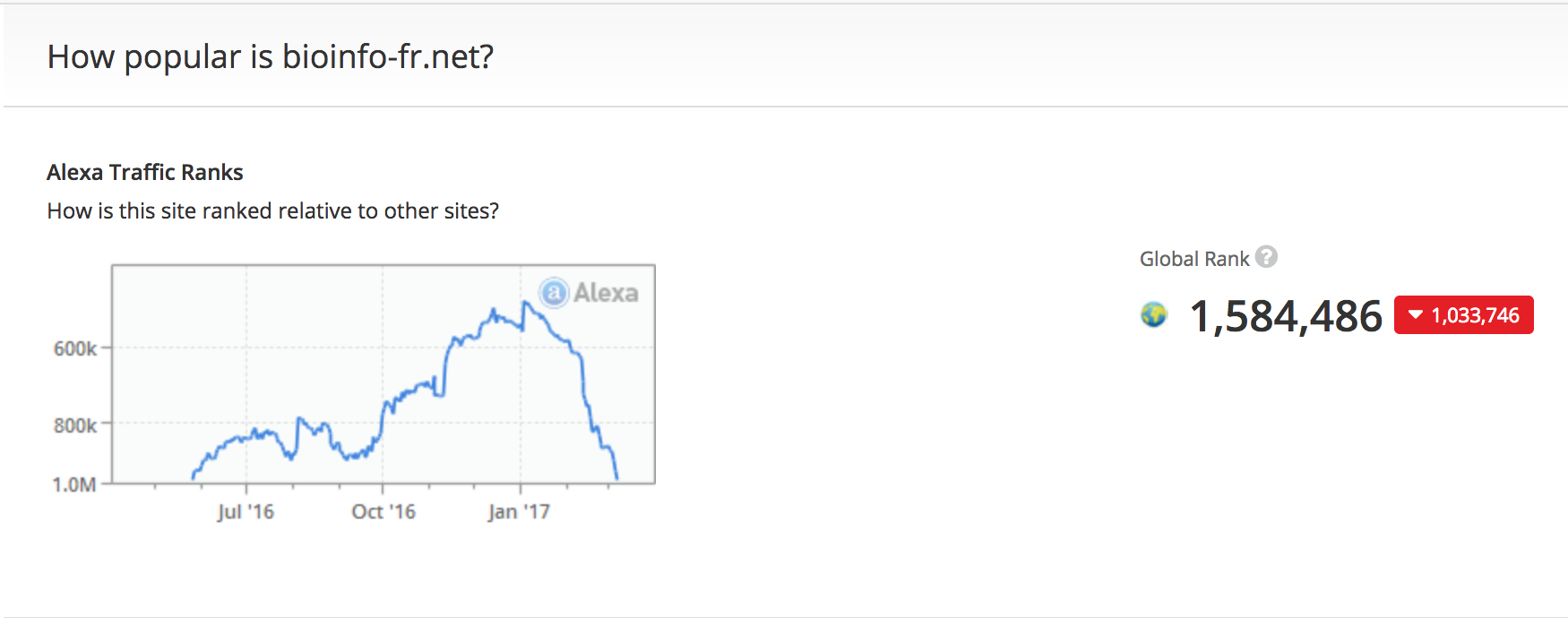
Avertissement : cet article déroge exceptionnellement à la ligne éditoriale que nous nous sommes imposées depuis le début de l'aventure. Nous n'allons pas parler de bioinformatique de près ou de loin dans cet article. Quoique les plus enthousiastes d'entre vous pourraient dire que cela peut arriver à une application web bioinfo 🙂 Mise en bouche Nous…
-
Comment fixer les problèmes de déploiement et de durabilité des outils en bioinformatique ? Indice : conda !
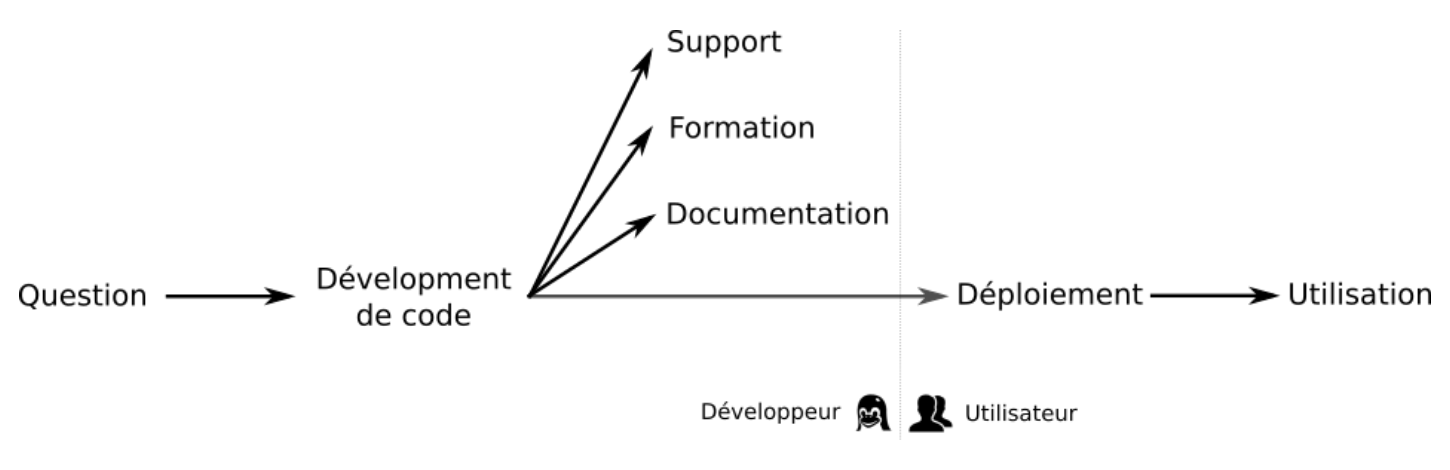
La diversité des questions que se posent nos amis biologistes entraîne une diversité des données : génomiques, images, etc. De plus, ces données sont générées à des vitesses folles. Pour manipuler les données et extraire les informations utiles, des solutions et outils bioinformatiques sont nécessaires. De nombreux outils existent déjà pour répondre à de nombreuses questions.…
-
Conda le meilleur ami du bioinformaticien

Conda, le meilleur ami du bioinformaticien Conda est un package manager écrit en python, comme pypi. Mais contrairement à celui-ci, il permet d'installer des programmes écrits dans d'autres langages. Notamment vos outils bioinformatiques préférés par l’intermédiaire du dépôt bioconda. C'est-à-dire que tous vos outils, que ce soit samtools, bwa, bowtie, trimmomatic, fastqc et j'en passe,…
-
Tour d'horizon des outils de visualisation des réseaux biologiques

Après les différentes méthodes d'analyse et de représentation de réseaux métaboliques biologiques, je vais vous parler des différents outils de visualisation. Car oui, la visualisation d'un réseau ou de ses sous-parties peut être le début de son analyse, car elle permet de se rendre compte de sa topologie, de sa complexité, sa connectivité… En biologie, on peut…
-
Dessiner vos gènes à partir d'un GFF3
Je découvre à l'instant genometools, un outil multifonction qui est capable de générer une image représentant des annotations sur un génome de référence. Idéal par exemple, si vous voulez vous passer d'un genome browser comme UCSC ou Ensembl pour incruster des images dans votre rapport. Installation Vous pouvez récupérer et compiler le code source depuis le site officiel. Pour…

