Étiquette : tutoriel
-
R : représenter des genomic tracks avec Gviz
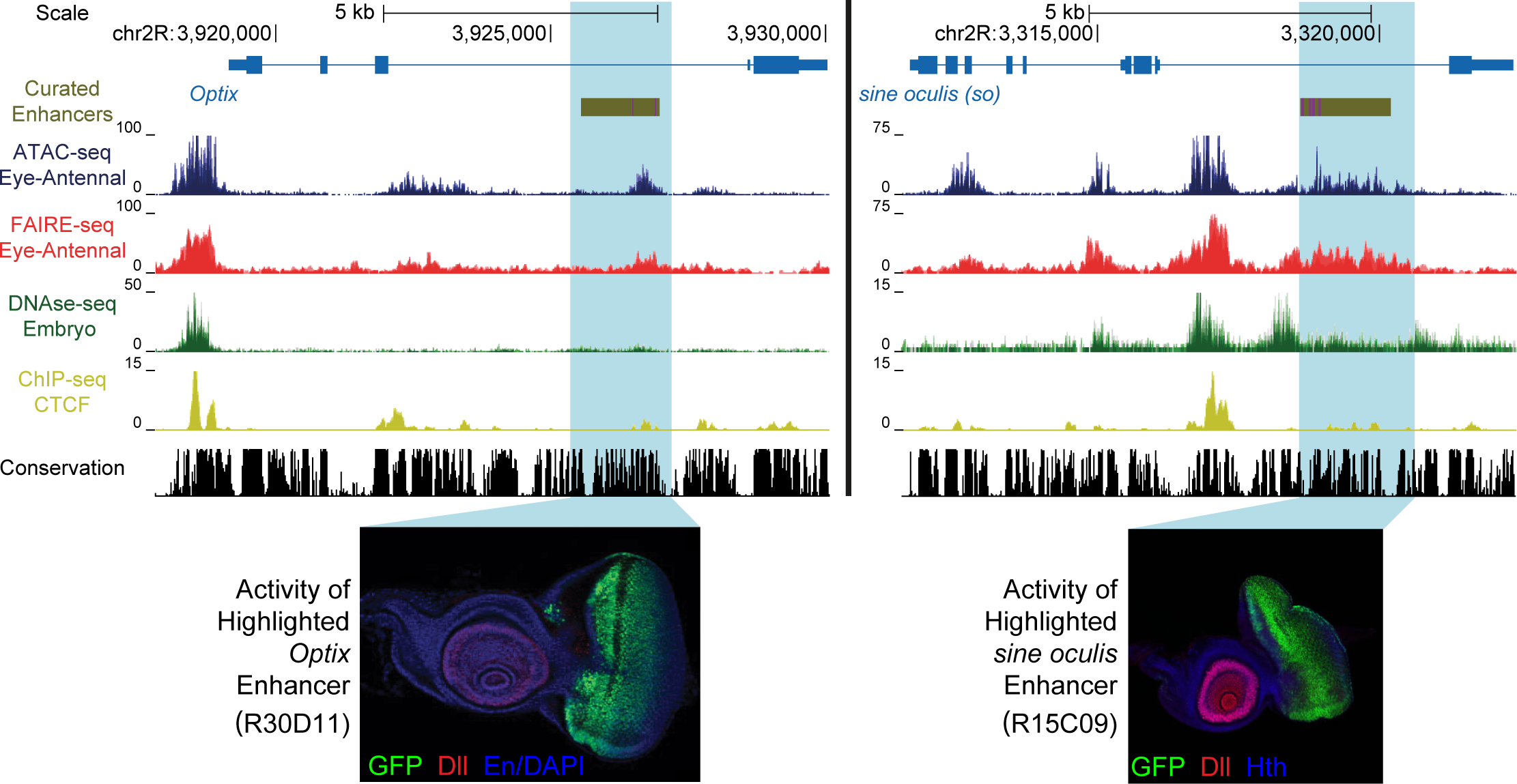
Si vous analysez des données d'épigénomique telles que de l'ATAC-seq ou des ChIP-seq, vous souhaitez sûrement pouvoir représenter des exemples de pics sous forme de genomic tracks comme on en voit souvent dans les publications. Lorsque l'on inspecte ses données de coverage (ou couverture), on charge généralement le fichier Bam ou le fichier BigWig dans…
-
Pourquoi je ne lis pas tous les CVs de la même manière

Dans un précédent billet je me suis laissé vagabonder autour de l'idée qu'un bioinformaticien était un data truc comme un autre. Dans ces lignes je vais exprimer pourquoi il est important à mon sens de bien travailler son CV… et ce qui personnellement me fait réfléchir sur la forme de CV liée à nos métiers.…
-
Qu'est-ce qu'un bon fichier Lisez-moi.txt

Vous venez de finir votre outil sur lequel vous travaillez depuis 1 semaine/1 mois/1 an/10 ans (rayez la mention inutile) qui va révolutionner votre domaine. Votre code est versionné, formaté, commenté, documenté, testé, les résultats sont évalués selon le gold standard de la discipline sur des jeux de données représentatifs de la réalité, votre publication…
-
Introduction à la manipulation d'intervalles dans R
Introduction "Quelle est la profondeur de ce séquençage ?" "Quelle proportion de SNPs se situent dans des exons ?" "Y a-t-il des pics dans ces données de ChIP-seq ?" "Quelle proportion de promoteurs chevauchent des îlots CpG ?" Voilà le genre de questions rencontrées fréquemment en bioinformatique. Nous pouvons y répondre à l'aide de la…
-
Installer JupyterHub pour des Notebooks hébergés sur votre serveur
Vous connaissez sans doute déjà les notebooks Jupyter [1], ces documents web où l'on peut rédiger du contenu en Markdown, pouvant contenir des formules mathématiques en LaTeX, mêlées à des cellules de code Python, (ou R, Julia etc.) que l'on peut exécuter au cas par cas de façon interactive. Ils sont pas mal utilisés en…
-
Pourquoi et comment déposer un package R sur Bioconductor ?

Ça y est, votre code R un poil brut commence à avoir de la substance et vous envisagez d'en faire un outil à part entière. Comme tout bioinformaticien qui se respecte, vous envisagez donc de packager (ou paqueter en français) proprement cet ensemble de scripts R. Non on ne largue pas une nuée de scripts…
-
Contrarié par les diagrammes de Venn ? Découvrez les diagrammes UpSet
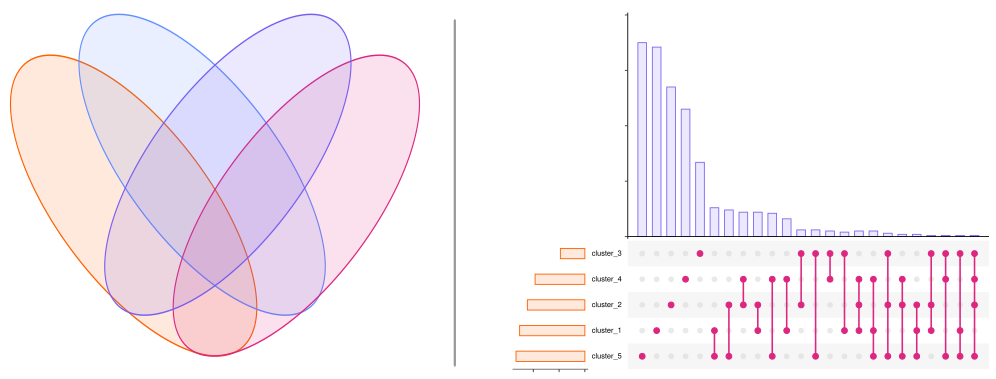
Titre incluant un moyen mnémotechnique ;D Avec ses cercles entrecroisés, on ne présente plus le célèbre diagramme de Venn. Cette représentation est utilisée dans le cas où l'on souhaite représenter le recoupement de données de nombre fini selon plusieurs variables qualitatives. De façon plus simple lorsqu'on a 2 variables qualitatives : combien d'éléments présents dans la…
-
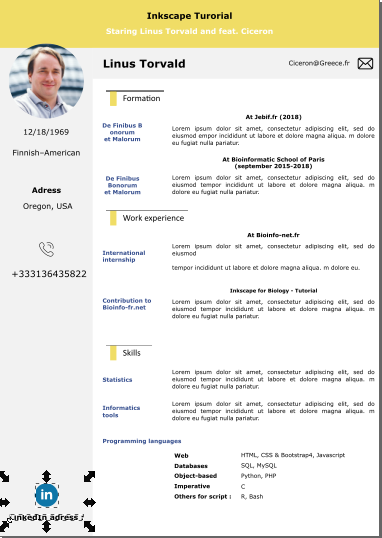
Du CV jusqu'au poster avec Inkscape (débutant)
Nous revoilà pour de nouvelles aventures sur Inkscape ! Le but de ce tuto est moins de faire son CV avec Inkscape, quand des outils qu’on utilise tous les jours le font très bien, que de se familiariser avec un outil puissant en manipulant des notions de bases qui peuvent servir ensuite notamment dans l’élaboration de…

