Catégorie : Découverte
-
Gérer les versions de vos fichiers : premiers pas avec git

Git est un logiciel de contrôle de versions de fichiers. Il est distribué sous licence GNU GPLv2 et est disponible sur les principaux systèmes d'exploitation. Cet article est le premier d'une série de deux. Nous allons voir ici (1) à quoi sert le contrôle de versions, (2) comment configurer git et (3) les bases de…
-
L'annotation de régions génomiques et les analyses d’enrichissement

Les annotations sont essentielles lors d'analyses fonctionnelles à large échelle sur le génome. Lorsque l’on pratique des analyses en génomique, basées sur des techniques comme le RNA-seq ou le ChIP-seq, on se retrouve avec respectivement une liste de transcrits ou de pics (régions génomiques). Dans le cas des analyses de ChIP-seq, on souhaite caractériser les gènes cibles du…
-
SARTools : l'analyse différentielle pour tous
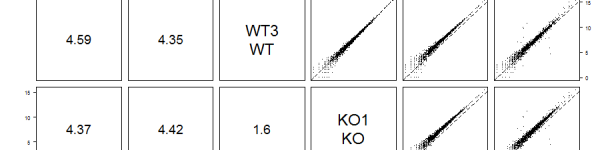
Un article court aujourd’hui pour présenter SARTools, un outil d’analyse différentielle de données de RNA-seq. Plus qu’un outil, SARTools est plutôt un pipeline simplifié pour traiter des données d’analyses réalisées dans un plan d’expérience assez basique. En effet le cadre d’utilisation de SARTools est volontairement limité aux plans d’expérience simples, à savoir des comparaisons de…
-
Former et se former en bioinformatique avec GOBLET !

Dans cet article, je vous présenterai un nouveau site qui peut être intéressant aussi bien pour les étudiants que pour les enseignants, voire également pour les bioinformaticiens déjà en fonction. Ici je ne vous noierai pas sous les lignes de code, mais je vous décrirai ce que le portail GOBLET vous propose en terme d'exercice.…
-
L'analyse en composantes principales (avec R)

L'ACP, ou Analyse en Composantes Principales, est une méthode d'exploration de données qui consiste à réduire la dimensionnalité du problème pour en extraire l'essentiel. Par une projection dans un espace plus petit, on réduit le nombre de variables, et si on réduit suffisamment on peut en faire un outil de diagnostic graphique. Comme c'est une…
-
Open Refine

Un grand coup de balai Le nettoyage de données est un défi en bioinformatique. Entre les personnes qui veulent réinventer les standards et les personnes qui ne savent pas les suivre, nous nous trouvons souvent en train de nettoyer, de formater et de changer la structure de nos données pour qu'elles soient conformes à une…
-
Python fait la numba

Suite à cet article, j'ai eu envie de comparer les temps d'exécution de Cython et Numba.Il est toujours intéressant de faire des tests de performances (benchmarks) de temps à autre, pour voir si on ne peut pas améliorer certains de nos algorithmes 🙂 Cython est un très bon moyen pour optimiser vos programmes, mais il…