Catégorie : Découverte
-
Les 10 ans du World Community Grid

Le 16 novembre 2014, le World Community Grid (WCG) fête ses 10 ans d’existence. Créé par IBM en 2004, ce projet de grille de calcul distribué par ordinateur se donne pour mission de soutenir des projets de recherche à fins humanitaires. Selon Wikipedia, une grille de calcul est une infrastructure virtuelle "qui exploite la puissance…
-
Comparaison de structures : le TM-Score

Pour comparer des structures 3D de protéines, nous avons vu le RMSD dans un précédent article. Je vous propose cette semaine de parler du TM-Score décrit par Zhang et Skolnick en 2004. Les bases Le TM-Score a été développé afin de calculer la qualité des structures de protéines prédites lors de la compétition CASP5. Le but…
-
Hi-C : Quelques bases
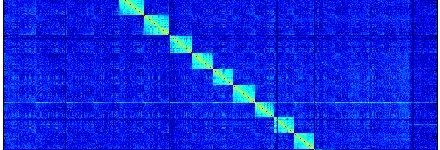
Aujourd'hui on va découvrir ensemble une des petites dernières dans la famille des techniques hauts débits : le High Chromosome Contact map (Hi C)[1] . Revenons sur quelques bases : un gène ne pourra être exprimé que si l'ADN qui le code est déplié. Par conséquent les régions dans lesquelles les gènes ne sont pas exprimés sont…
-
École Polytechnique Fédérale de Lausanne (EPFL)
Il y a quelque temps, Akira vous présentait l'EMBL en Allemagne. Place maintenant à la Suisse et à sa mythique EPFL, lieu où vous pourrez espérer décrocher un stage ou même poser vos bagages pour un contrat un peu plus long. Histoire et géographie Pour commencer, L'EPFL ne se situe pas tout à fait comme…
-
Le rôle de la bioinformatique dans les labos collaboratifs

Y a-t-il une place pour la bioinformatique dans les structures du type « Do it yourself biology », « Bio-hackers spaces » et autres FabLabs ? Avant de répondre à cette question, quelques précisions sur ces espaces collaboratifs sont nécessaires. Il s’agit de curieux et de passionnés qui se retrouvent dans des caves… ou des laboratoires pour élaborer des projets…
-
Comparaison de structures : le RMSD

En 1973, Anfinsen montrait qu'une protéine se replie en une structure unique et stable. Même si des exceptions existent, cette règle s'applique à la plupart des petites protéines globulaires. La structure des protéines est non seulement stable mais aussi plus conservée que la séquence au cours de l'évolution. En effet, des protéines ayant des séquences très…
-
Cython : votre programme Python mais 100x plus vite

Python est un langage extrêmement pratique car il est facile à lire et à écrire, comparé à un langage de "bas niveau" et compilé comme le C. D'un autre côté, à l'exécution il est beaucoup plus lent. C'est un compromis entre les deux qu'offre Cython, permettant d'accélérer votre programme d'un facteur 2 à plus de…
-
Introduction aux systèmes multi-agents

Dans le monde merveilleux de l'intelligence artificielle, il existe une catégorie de systèmes de simulation inspirés de la nature et particulièrement adaptés à de nombreuses problématiques en biologie : les systèmes multi-agents. Ces systèmes sont composés d'entités autonomes (les agents) qui interagissent entre elles et avec un environnement dans lequel elles évoluent. Bien évidemment, l'inspiration "biologique"…

