Étiquette : tutoriel
-
Automatiser la récupération de données biologiques
Qui dit bioinformatique, dit récupération et manipulation des données. Ces données peuvent être générées à partir d'algorithmes ou bien récupérées depuis des bases de données biologiques. Aujourd'hui, le nombre de ces bases de données est en constante augmentation. Chaque mécanisme biologique, famille moléculaire ou organisme est associé à un ou plusieurs de ces dépôts de données. Si un…
-
Snakemake pour les nuls (ou comment créer un pipeline facilement ?)

Bonjour à tous, et bienvenue dans le premier épisode d'une (longue ?) série de prise en main de l'outil dédié au pipeline : Snakemake. Si vous ne connaissez pas encore cet outil, c'est que vous êtes sûrement passés à côté de cet article écrit par Nisaea. Alors, quel sera les bénéfices de retranscrire vos pipelines déjà…
-
Comment faire de Sozi présentations avec inkscape
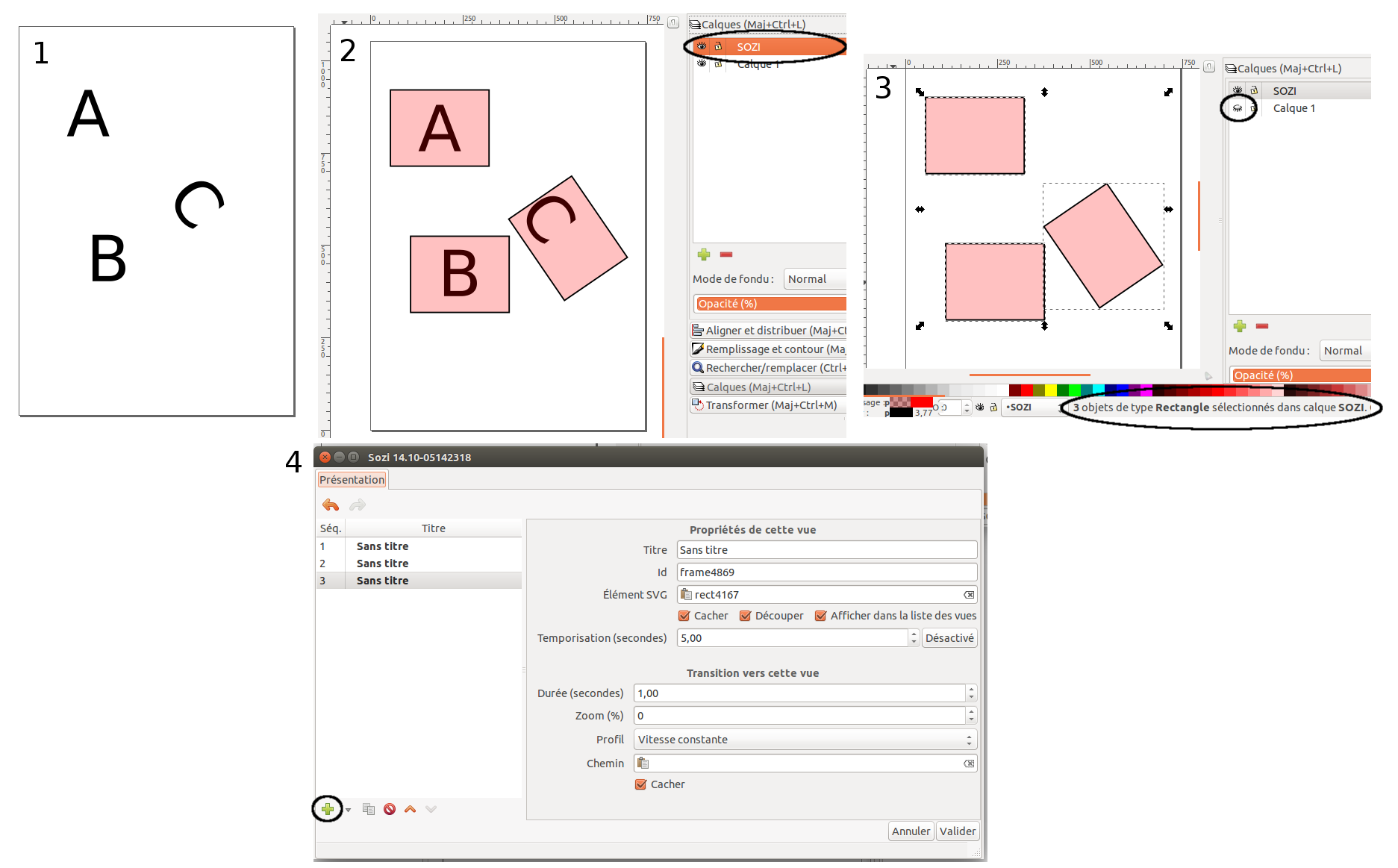
Vous avez sûrement vu le billet sur inkscape écrit par Zazo0o. Maintenant que vous avez franchi le pas et que vous maîtrisez cet outil, nous allons voir qu'il est également possible de créer des présentations dynamiques grâce au plugin Sozi (qui est en plus développé par des français "cocorico"). Les auteurs ont arrêté le développement…
-
Python, dessine-moi un graphe
Derrière ce titre énigmatique, qui n'aura pas été sans vous rappeler la fameuse phrase du Petit Prince d'Antoine de Saint Exupéry, se cache un module pour Python qui dira sûrement quelque chose à nos lecteurs assidus spécialisés dans les graphes : pygraphviz ! Ce module a été créé autour de GraphViz et vous permet ainsi de faire…
-
Twitter, arme de communication massive et outil scientifique

Twitter est un réseau social incontournable. Son principe est simple, il s'agit de s'échanger avec ses "followers", des messages ou "tweets" de 140 charactères maximum. Il est surtout utilisé aux Etats-Unis, mais la fièvre Twitter gagne peu à peu l'Europe. De nombreux scientifiques, chefs de labos, doctorants et même des institutions informent par ce biais. En créant…
-
De la procrastination dans l'R

Connaissances requises Introduction Si l'on s'en réfère à la définition : Un informaticien, et a fortiori un bioinformaticien, fera tout pour mettre en œuvre des stratégies lui permettant d'automatiser les tâches répétitives qui lui incombe. Plusieurs avantages à cela, i/ rallonger les pauses café, ii/ profiter du temps gagné pour regarder la guerre des étoiles directement…
-
L'annotation de régions génomiques et les analyses d’enrichissement

Les annotations sont essentielles lors d'analyses fonctionnelles à large échelle sur le génome. Lorsque l’on pratique des analyses en génomique, basées sur des techniques comme le RNA-seq ou le ChIP-seq, on se retrouve avec respectivement une liste de transcrits ou de pics (régions génomiques). Dans le cas des analyses de ChIP-seq, on souhaite caractériser les gènes cibles du…
-
Former et se former en bioinformatique avec GOBLET !

Dans cet article, je vous présenterai un nouveau site qui peut être intéressant aussi bien pour les étudiants que pour les enseignants, voire également pour les bioinformaticiens déjà en fonction. Ici je ne vous noierai pas sous les lignes de code, mais je vous décrirai ce que le portail GOBLET vous propose en terme d'exercice.…

