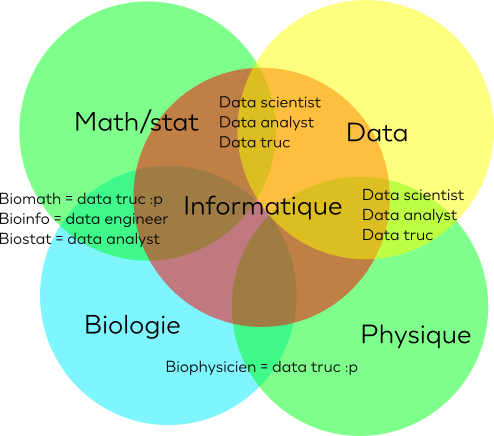
Qui sommes nous ?
-
Licence Bio-Informatique, Statistique et Modélisation (BISM) à Lyon 1
 Lire la suite : Licence Bio-Informatique, Statistique et Modélisation (BISM) à Lyon 1
Lire la suite : Licence Bio-Informatique, Statistique et Modélisation (BISM) à Lyon 1Cet article est une mise à jour de https://bioinfo-fr.net/presentation-de-la-licence-miv-a-lyon-1. Cette formation pluridisciplinaire existe depuis 2010 (ex. Modélisation et Informatique du Vivant), et bénéficie de la recherche historique lyonnaise en analyses de données biologiques et notamment en biométrie et statistiques. Les aspects liés à la bioinformatique ont été renforcés à partir de 2018, en lien avec […]
-
Alignement de séquences : bien comprendre l'incontournable algorithme de Needleman-Wunsch
 Lire la suite : Alignement de séquences : bien comprendre l'incontournable algorithme de Needleman-Wunsch
Lire la suite : Alignement de séquences : bien comprendre l'incontournable algorithme de Needleman-WunschIntroduction L'analyse des séquences biologiques (ADN, ARN ou protéines) est un sujet central de la biologie moléculaire et de la bioinformatique. Dans ce cadre, chercher à savoir à quel point deux séquences sont similaires ou dissimilaires est une des questions qu'on se pose le plus souvent (par exemple dans le but d'analyser des relations phylogénétiques). […]
-
Des molécules et des données !
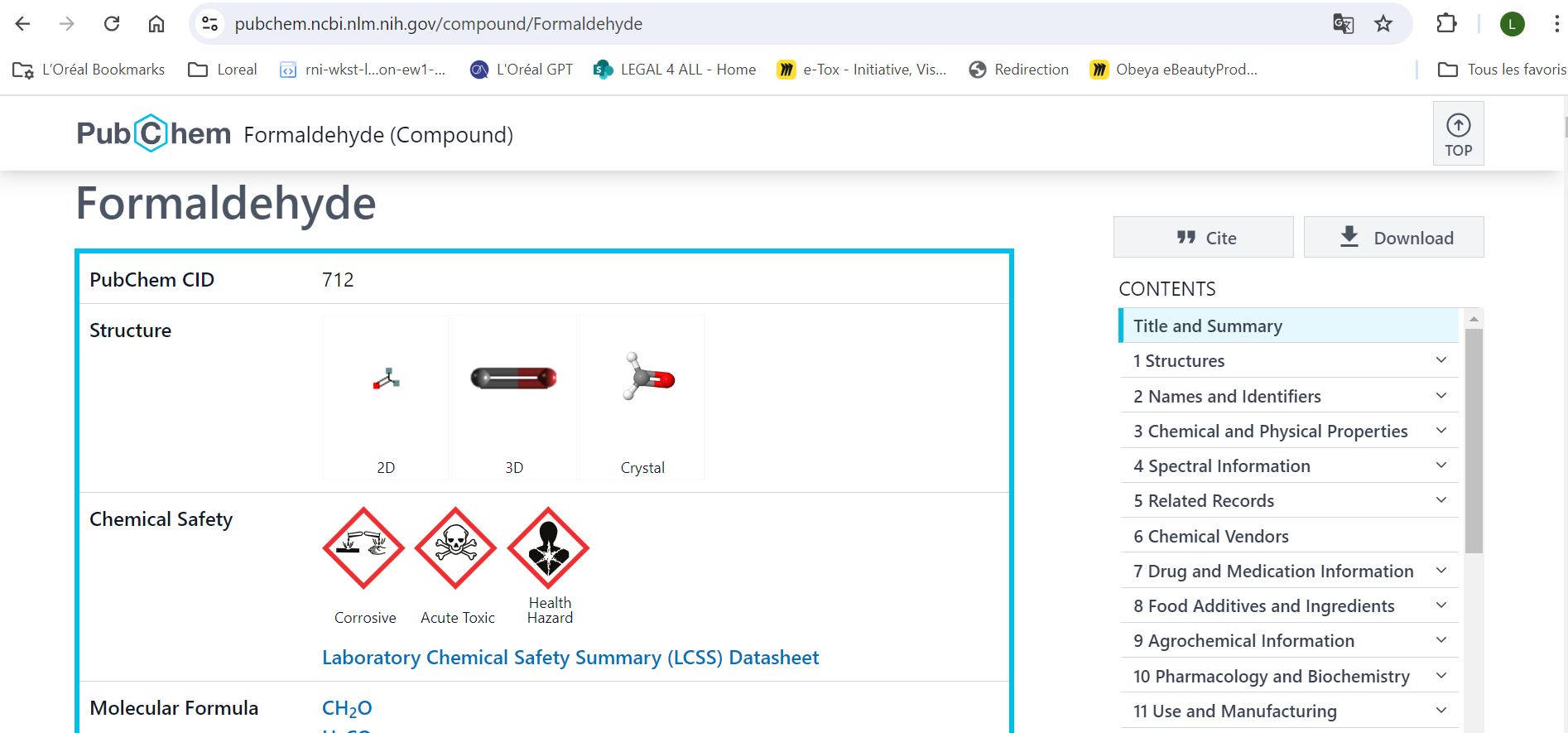 Lire la suite : Des molécules et des données !
Lire la suite : Des molécules et des données !Vu que nous sommes sur un blog de bio-informatique, je vous propose aujourd'hui de parler chimie ! Enfin plutôt, chémoinformatique* et quelques notions de la façon dont une molécule est stockée en ligne en fonction des réglementations chimiques et cosmétiques. Je ne suis pas un expert de ce domaine, mais alors vraiment pas du tout. Par […]
-
Software Heritage : l'archive universelle des codes sources logiciels
 Lire la suite : Software Heritage : l'archive universelle des codes sources logiciels
Lire la suite : Software Heritage : l'archive universelle des codes sources logicielsDans cet article, je vous propose de découvrir Software Heritage, l’archive universelle dédiée à la collecte, la préservation et le partage des codes sources logiciels. En 2021, je suis devenu ambassadeur pour Software Heritage, c’est-à-dire représentant de la communauté bioinformatique, dans l’infrastructure Software Heritage. Pourquoi archiver son code source ? Aujourd’hui, les scientifiques utilisent très souvent des […]
-
Conda et le piège de la licence Anaconda
 Lire la suite : Conda et le piège de la licence Anaconda
Lire la suite : Conda et le piège de la licence AnacondaConda est apparu en 2012 et a été distribué par Continuum Analytics, connue sous le nom d’Anaconda Inc depuis 2017. Conda est distribué sous la licence MIT, l’une des licences open-source les plus permissives, permettant aux utilisateurs d’adopter, d’adapter et de partager le logiciel presque sans restrictions. Conda a été conçu pour gérer les environnements […]
-
ABSD : base de données d'anticorps non redondants et standardisés
 Lire la suite : ABSD : base de données d'anticorps non redondants et standardisés
Lire la suite : ABSD : base de données d'anticorps non redondants et standardisésLes anticorps (immunoglobulines) jouent un rôle crucial dans la réponse immunitaire contre les menaces extérieures, telles les infections virales. Une immunoglobuline est composée de deux molécules en interaction appelées chaîne légère et chaîne lourde : la combinaison d’une chaîne légère et d’une chaîne lourde donne une immunoglobuline (voir Figure 1). Bien que le nombre théorique d'immunoglobulines […]
-
Comment l'étude des intensités d'expression de gène a évolué avec les technologies de transcriptomique ?
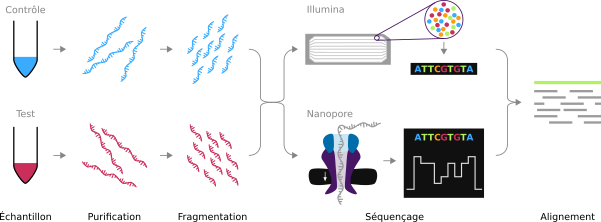 Lire la suite : Comment l'étude des intensités d'expression de gène a évolué avec les technologies de transcriptomique ?
Lire la suite : Comment l'étude des intensités d'expression de gène a évolué avec les technologies de transcriptomique ?Cet article est en partie basé sur mon introduction de thèse [0], reformatée pour convenir au format du blog et agrémentée des nouvelles informations pour ne pas être un simple catalogue de connaissances. Qu'il s'agisse du génome entier, du génome de tissus ou du génome de cellules spécifiques, l'étude de la quantité de transcrits produits, […]
-
Édito : Bioinfo-fr vous souhaite la bonne année 2025
 Lire la suite : Édito : Bioinfo-fr vous souhaite la bonne année 2025
Lire la suite : Édito : Bioinfo-fr vous souhaite la bonne année 2025Chers lecteurs, chères lectrices, la nouvelle année commence, et vous avez peut-être (ou pas) pris de bonnes résolutions. La nôtre est de vous offrir la meilleure plateforme de communication possible autours de la bioinfo. Bioinfo-fr existe pour vous, et vie grâce à vous, et nous vous en sommes profondément reconnaissants. Nous essayons de vous fournir […]
Articles les plus lus ces 7 derniers jours
Les salons de discussion :



La boutique

Quelques liens
Étiquettes
ADN analyse base de données bioinformatique code concours conférence débutant Découverte edito emploi formation Génomique Interview JeBiF JOBIM langage master outil programmation Python R Recherche rentrée script SFBI statistiques séquençage tutoriel vacances visualisation