Qui sommes nous ?
-
Conda et le piège de la licence Anaconda
 Lire la suite : Conda et le piège de la licence Anaconda
Lire la suite : Conda et le piège de la licence AnacondaConda est apparu en 2012 et a été distribué par Continuum Analytics, connue sous le nom d’Anaconda Inc depuis 2017. Conda est distribué sous la licence MIT, l’une des licences open-source les plus permissives, permettant aux utilisateurs d’adopter, d’adapter et de partager le logiciel presque sans restrictions. Conda a été conçu pour gérer les environnements […]
-
ABSD : base de données d'anticorps non redondants et standardisés
 Lire la suite : ABSD : base de données d'anticorps non redondants et standardisés
Lire la suite : ABSD : base de données d'anticorps non redondants et standardisésLes anticorps (immunoglobulines) jouent un rôle crucial dans la réponse immunitaire contre les menaces extérieures, telles les infections virales. Une immunoglobuline est composée de deux molécules en interaction appelées chaîne légère et chaîne lourde : la combinaison d’une chaîne légère et d’une chaîne lourde donne une immunoglobuline (voir Figure 1). Bien que le nombre théorique d'immunoglobulines […]
-
Comment l'étude des intensités d'expression de gène a évolué avec les technologies de transcriptomique ?
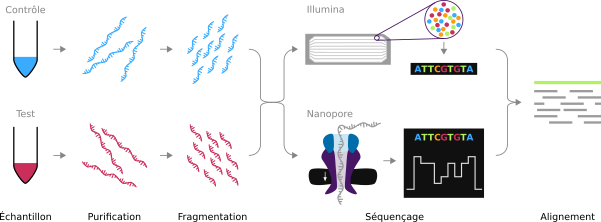 Lire la suite : Comment l'étude des intensités d'expression de gène a évolué avec les technologies de transcriptomique ?
Lire la suite : Comment l'étude des intensités d'expression de gène a évolué avec les technologies de transcriptomique ?Cet article est en partie basé sur mon introduction de thèse [0], reformatée pour convenir au format du blog et agrémentée des nouvelles informations pour ne pas être un simple catalogue de connaissances. Qu'il s'agisse du génome entier, du génome de tissus ou du génome de cellules spécifiques, l'étude de la quantité de transcrits produits, […]
-
Édito : Bioinfo-fr vous souhaite la bonne année 2025
 Lire la suite : Édito : Bioinfo-fr vous souhaite la bonne année 2025
Lire la suite : Édito : Bioinfo-fr vous souhaite la bonne année 2025Chers lecteurs, chères lectrices, la nouvelle année commence, et vous avez peut-être (ou pas) pris de bonnes résolutions. La nôtre est de vous offrir la meilleure plateforme de communication possible autours de la bioinfo. Bioinfo-fr existe pour vous, et vie grâce à vous, et nous vous en sommes profondément reconnaissants. Nous essayons de vous fournir […]
-
Les Bioinformations n°40 sont là !
 Lire la suite : Les Bioinformations n°40 sont là !
Lire la suite : Les Bioinformations n°40 sont là !Quelles sont les dernières actualités des communautés françaises de bioinformatique ? Vous saurez tout dans ce nouveau numéro des Bioinformations : https://www.bioinformations.fr C'est quoi déjà les Bioinformations ? Il s'agit d'une newsletter trimestrielle créée en 2012, à l'initiative de la Société Française de Bioinformatique (SFBI), l'association de Jeunes Bioinformaticiens de France (JeBiF) et nous-mêmes, le blog collaboratif Bioinfo-fr.net, […]
-
Chronique d'une soumission de données à GEO
 Lire la suite : Chronique d'une soumission de données à GEO
Lire la suite : Chronique d'une soumission de données à GEOJe vais vous raconter étape par étape ma soumission de données de séquençage à la base de données de génomique GEO (Gene Expression Omnibus) d'un projet en cours de finition. Sommaire GEO, Qu'est-ce que c'est ? Lorsque l'on veut publier les résultats d'une étude comprenant du séquençage haut débit, nous devons publier les données brutes et […]
-
Concours CRCN-CNRS 2025
 Lire la suite : Concours CRCN-CNRS 2025
Lire la suite : Concours CRCN-CNRS 2025Comme tous les ans, le CNRS ouvre de nouveaux postes permanents de chargé de recherche au concours, et comme chaque année, la communication autours du concours est une vraie nébuleuse. D'après l'INS2I (branche sciences informatiques du CNRS), la campagne 2025 devrait s'ouvrir ce 9 décembre et se clôturer le 8 janvier 2025 : https://www.ins2i.cnrs.fr/fr/cnrsinfo/concours-chercheurs-2025-liste-des-postes-abondes-par-cnrs-sciences-informatiques Il s'agit […]
-
Les pièges mentaux à éviter pour les plus jeunes chercheurs
 Lire la suite : Les pièges mentaux à éviter pour les plus jeunes chercheurs
Lire la suite : Les pièges mentaux à éviter pour les plus jeunes chercheursAujourd'hui, j'aimerais exprimer une opinion que je ne vois pas et qui fait suite à quelques conseils données aux doctorants dans ce blog. Ce message s'adresse à tous, dans une communauté à laquelle je n'appartiens plus vraiment n'étant plus dans l'académique. Nous évoquerons ici certains clichés que j'ai entendu. Un jour on est jeune, on […]
Articles les plus lus ces 7 derniers jours
Les salons de discussion :



La boutique

Quelques liens
Étiquettes
ADN analyse base de données bioinformatique code concours conférence débutant Découverte edito emploi formation Génomique Interview JeBiF JOBIM langage master outil programmation Python R Recherche rentrée script SFBI statistiques séquençage tutoriel vacances visualisation







