Qui sommes nous ?
-
De la procrastination dans l'R
 Lire la suite : De la procrastination dans l'R
Lire la suite : De la procrastination dans l'RConnaissances requises Introduction Si l'on s'en réfère à la définition : Un informaticien, et a fortiori un bioinformaticien, fera tout pour mettre en œuvre des stratégies lui permettant d'automatiser les tâches répétitives qui lui incombe. Plusieurs avantages à cela, i/ rallonger les pauses café, ii/ profiter du temps gagné pour regarder la guerre des étoiles directement […]
-
Présentation du Master Bioinformatique & Génomique de Rennes
 Lire la suite : Présentation du Master Bioinformatique & Génomique de Rennes
Lire la suite : Présentation du Master Bioinformatique & Génomique de RennesPrésentation Le Master Bioinformatique & Génomique (BIG) de l'Université de Rennes 1 a pris en 2013 la suite du Master Modélisation de Systèmes Biologiques (MSB). L’objectif principal de ce Master est de former des étudiants issus d’un cursus biologique, informatique ou mathématique aux besoins pluridisciplinaires en bio-informatique et en technologies à haut-débit de génomique fonctionnelle et […]
-
Automatiser le parcours et la manipulation d’arbres phylogénétiques avec le module Bio.Phylo de BioPython
 Lire la suite : Automatiser le parcours et la manipulation d’arbres phylogénétiques avec le module Bio.Phylo de BioPython
Lire la suite : Automatiser le parcours et la manipulation d’arbres phylogénétiques avec le module Bio.Phylo de BioPythonLa génomique comparative permet d'étudier l'évolution d'organismes par comparaison de leur génome. La représentation de la proximité entre les organismes, élément essentiel de la génomique comparative, repose sur des arbres phylogénétiques. Mais comment manipuler ces arbres facilement ? Quand il n’y en a qu’un, pas de problème : on utilise un visualisateur comme ceux proposés dans l’article sur les […]
-
L'annotation de régions génomiques et les analyses d’enrichissement
 Lire la suite : L'annotation de régions génomiques et les analyses d’enrichissement
Lire la suite : L'annotation de régions génomiques et les analyses d’enrichissementLes annotations sont essentielles lors d'analyses fonctionnelles à large échelle sur le génome. Lorsque l’on pratique des analyses en génomique, basées sur des techniques comme le RNA-seq ou le ChIP-seq, on se retrouve avec respectivement une liste de transcrits ou de pics (régions génomiques). Dans le cas des analyses de ChIP-seq, on souhaite caractériser les gènes cibles du […]
-
RNA-seq : plus de profondeur ou plus d'échantillons ?
 Lire la suite : RNA-seq : plus de profondeur ou plus d'échantillons ?
Lire la suite : RNA-seq : plus de profondeur ou plus d'échantillons ?Lorsque l'on se lance dans l'aventure du séquençage haut débit de transcriptome, on est amené à se poser LA question, oui LA, celle que l'on redoute à peu près tous quand on a un budget serré : À quelle profondeur dois-je séquencer mes échantillons ? Toutes les publications s'accordent à le dire, plus on a de réplicats, […]
-
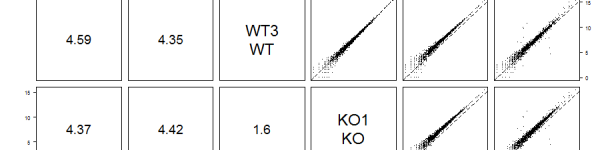
SARTools : l'analyse différentielle pour tous
 Lire la suite : SARTools : l'analyse différentielle pour tous
Lire la suite : SARTools : l'analyse différentielle pour tousUn article court aujourd’hui pour présenter SARTools, un outil d’analyse différentielle de données de RNA-seq. Plus qu’un outil, SARTools est plutôt un pipeline simplifié pour traiter des données d’analyses réalisées dans un plan d’expérience assez basique. En effet le cadre d’utilisation de SARTools est volontairement limité aux plans d’expérience simples, à savoir des comparaisons de […]
-
Ecole doctorale Française en Bio-informatique : Les dates à ne pas manquer
 Lire la suite : Ecole doctorale Française en Bio-informatique : Les dates à ne pas manquer
Lire la suite : Ecole doctorale Française en Bio-informatique : Les dates à ne pas manquerEn France, pour faire une thèse, il faut dans la plus grande partie des cas candidater à une école doctorale. Ces écoles, souvent assignées à une ville ou à une région, ont chacune leurs propres emplois du temps et leurs propres règles. Nous vous proposons ici une liste des dates à ne pas manquer pour […]
Articles les plus lus ces 7 derniers jours
Les salons de discussion :



La boutique

Quelques liens
Étiquettes
ADN analyse base de données bioinformatique code concours conférence débutant Découverte edito emploi formation Génomique Interview JeBiF JOBIM langage master outil programmation Python R Recherche rentrée script SFBI statistiques séquençage tutoriel vacances visualisation