Qui sommes nous ?
-
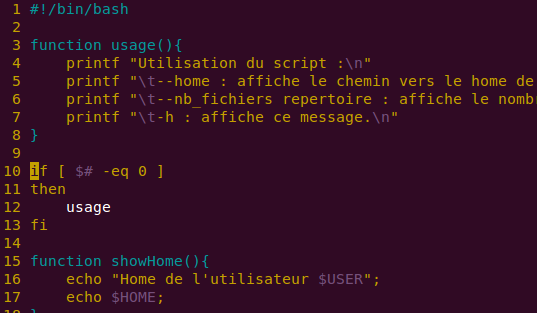
LaTeX : les flottants !
 Lire la suite : LaTeX : les flottants !
Lire la suite : LaTeX : les flottants !Chose promise, chose due dans le précédent article voilà les flottants dans LaTeX Les flottants sont l'ensemble des éléments qui perturbent le flot du texte (pour faire simple) et désignent classiquement les tables et les figures ;on peut définir des environnements custom de flottants, mais on y reviendra dans un autre article ;). Les flottants « nagent » dans le […]
-
2017, on y est !
 Lire la suite : 2017, on y est !
Lire la suite : 2017, on y est !L'ensemble de l'équipe de bioinfo-fr.net vous souhaite une excellente année 2017 et plein de réussites pro et perso (oui, on a attendu le dernier jour de janvier pour vous dire ça 🙂 ). Nous avons prévu quelques nouveautés à venir dans le courant de l'année. Nous essayons de faire au mieux et au plus rapide […]
-
dplyr et le génome humain
Lire la suite : dplyr et le génome humainIntroduction Non, ne fuyez pas tout de suite, chers lecteurs, tout va s'éclaircir : dplyr, c’est plyr pour les data.frame (les tableaux de données). Attendez, j’y viens, plyr, c’est un package R pour appliquer (apply) des fonctions. Donc, dplyr (prononcez “diplir”), c’est un package R, pour appliquer des fonctions à un tableau de données. Et ça, […]
-
Nowel approche…
Lire la suite : Nowel approche…… et avec lui son lot de festivités et festins en tout genre à venir ! Le blog s'est encore enrichi cette année de nombreux et beaux articles, et ce grâce à la participation de tous nos auteur.e.s et relecteur.trice.s toujours au top ! Merci à eux pour leur travail. Et si jamais vous, oui vous qui […]
-
La magie de LXD
 Lire la suite : La magie de LXD
Lire la suite : La magie de LXDÉcrire un pipeline en bioinformatique, c'est bien ! Le rendre portable c'est encore mieux ! Les bioinformaticiens oublient souvent ce dernier point et rare sont les pipelines qui marchent du premier coup. À vrai dire les circonstances ne sont pas en leur faveur. Un pipeline c'est plein de dépendances d'applications dans telle ou telle version, qu'il faut […]
-
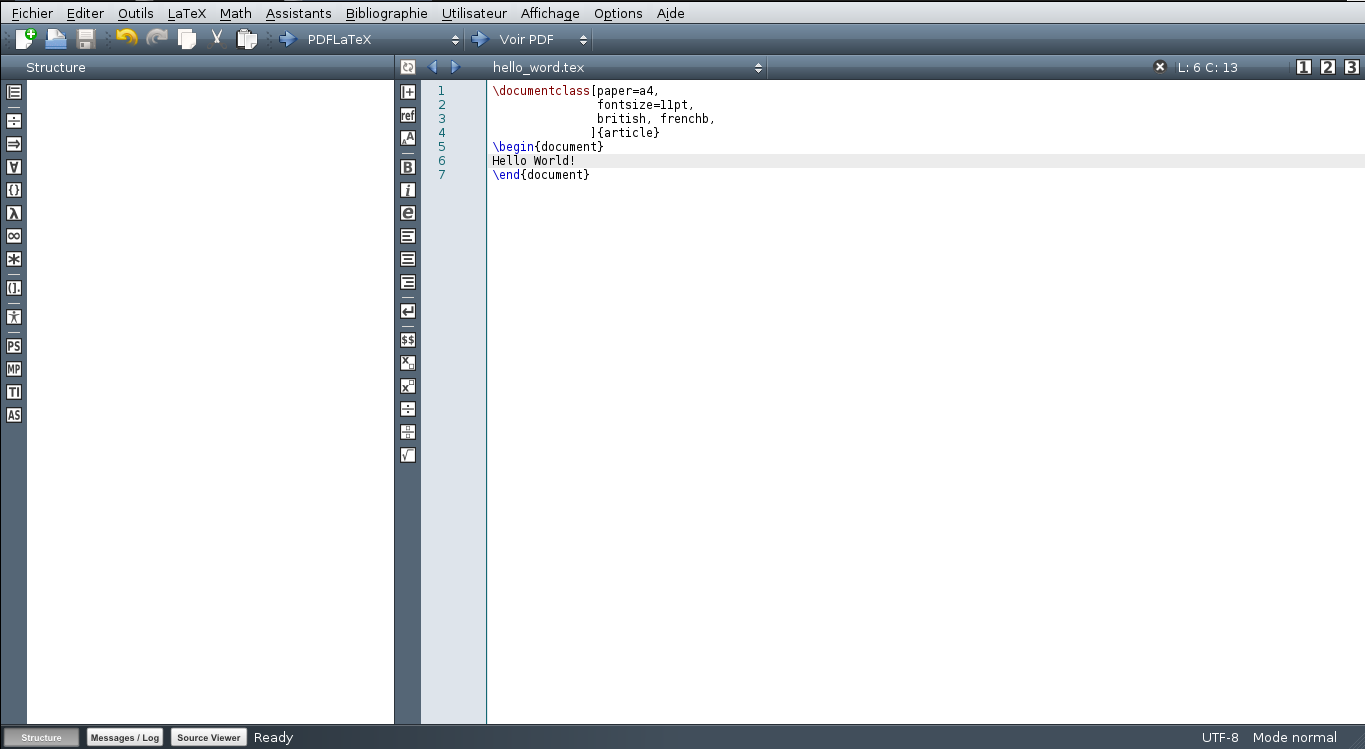
LaTeX : le premier document !
 Lire la suite : LaTeX : le premier document !
Lire la suite : LaTeX : le premier document !On s'est rendu compte sur #bioinfo-fr et sur #jebif (les canaux IRC sur freenode) qu'au cours des rédactions de rapports de stage ou même de thèse on avait plein de questions portant sur LaTeX et on a décidé qu'on allait se fendre de quelques articles sur LaTeX pour avoir une suite de liens appropriés à […]
-
Automatiser la récupération de données biologiques : version avancée
 Lire la suite : Automatiser la récupération de données biologiques : version avancée
Lire la suite : Automatiser la récupération de données biologiques : version avancéeDans cette version avancée, nous allons nous intéresser à une autre manière de récupérer des données depuis un site internet : via l'utilisation de formulaires. Les formulaires, ce sont ces pages avec plein de champs à remplir et que vous soumettez ensuite pour vous créer un compte, contacter votre hotline ou que sais-je. Leur utilisation permet […]
-
AMBB : Colloque « Les futurs de la bio-informatique »
 Lire la suite : AMBB : Colloque « Les futurs de la bio-informatique »
Lire la suite : AMBB : Colloque « Les futurs de la bio-informatique »L’Association du Master Bio-informatique de Bordeaux (AMBB) organise son premier colloque sur la thématique : « Les futurs de la bio-informatique ». Il aura lieu Jeudi 08 Décembre 2016 de 13H à 17H30 dans l’amphithéâtre B du bâtiment A29 sur le campus de Talence de l’Université de Bordeaux (351 Cours de la Libération, 33800 Talence). Cette demi-journée a […]
Articles les plus lus ces 7 derniers jours
Les salons de discussion :



La boutique

Quelques liens
Étiquettes
ADN analyse base de données bioinformatique code concours conférence débutant Découverte edito emploi formation Génomique Interview JeBiF JOBIM langage master outil programmation Python R Recherche rentrée script SFBI statistiques séquençage tutoriel vacances visualisation